|
乙内酰脲潜在多晶型的氢键分析背景 有机物分子晶体中的多晶型既为固化应用、制药开发以及精细和特种化学品行业带来了挑战,也带来了机遇。分子排列的不同会极大影响其性质,例如溶解度、生物利用度和稳定性等。表征和分类堆积方式差异的一种方法是分析氢键拓扑结构或基序。图1给出了一些常见类型氢键基序的示意图。 在Materials Studio中,基序被标注为nM[s],其中M标记基序类型,n是M该类型基序的数量,s是M类型基序的尺寸。CCDC区分以下基序类型,M:
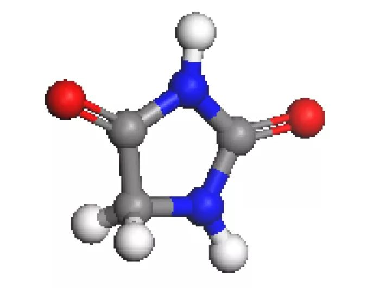
图1 一些常见氢键基序的示意图 R=环 C=无限链 DC=离散链 S=分子内接触 M=分子间接触 本教程讨论由Polymorph计算生成基序信息所需的基本步骤。首先,分子以代表其结构关键特征的官能团来表征。接下来,氢键施主和受主被分配到这些官能团中。当已准备分析分子时,CCDC模块从CSD数据库查询包含已定义官能团的化合物。根据它们的键序信息分析结果的匹配度。最后,分析Polymorph计算的结果,进行评分,并将每个推荐多晶型的基序信息与CSD查询的结果进行比较。 注:基序使用CCDC的CSD(Allen et al., 2002)和Mercury(McRae et al., 2006)功能。这些工具可直接从CCDC获得。 介绍 乙内酰脲是第三次CCDC多晶型预测盲测实验中使用的分子之一(Day et al., 2005)。当使用Polymorph预测乙内酰脲的堆积方式时,将预测实验观察到的晶体结构以及另外两种具有较低晶格能的结构。在本教程中,将使用CCDC基序搜索CCDC Motif Search生成预测氢键基序与已知结构接近程度的统计度量。 本教程包括如下部分: 开始 绘制乙内酰脲分子结构 准备结构并定义官能团和施主/受主位点 利用CSD查询相关结构 为Polymorph输出分配基序信息,并评分排名靠前的Polymorph结构 注意:为了和本教程中的参数保持一致,可以使用Settings Organizer对话框将项目中所有参数都设置为BIOVIA的默认值。 1、开始 首先启动Materials Studio并创建一个新项目。 打开New Project对话框,输入hydantoin作为项目名,单击OK按钮。 新项目将以hydantoin为项目名显示于Project Explorer中。 2、绘制乙内酰脲分子结构 在Project Explorer,右键单击工程根目录,并选择New | 3D Atomistic Document。 将新建的结构文档命名为hydantoin-mol。使用绘图工具绘制一个乙内酰脲分子(如下图所示),并通过单击Clean按钮对结构进行初步整理。 3、准备结构并定义官能团和施主/受主位点 在本节中,将使用官能团Functional Groups对话框定义官能团和施主/受主位点。在利用CSD进行查询时,官能团对于定义类似结构的组成至关重要。 从菜单栏中选择Modules | CCDC | Functional Groups,或单击Modules工具栏上的 按钮并选择Functional Groups,以打开Functional Groups对话框。
乙内酰脲分子包含两个等效的施主和受主位点。要运行有意义的CCDC基序搜索,应该选择一个同时包含施主(氧)和受主(氮)位点的官能团。满足这一要求的最小官能团是O=C-N,然而O=C-NH-C片段包含更多的分子拓扑结构。也可以选择整个分子作为官能团。定义的官能团类型将影响从CSD检索的结果,如何定义官能团取决于将如何应用搜索结果。在本教程中,将尝试检索尽可能多的相关结构,以最大化评分函数的统计相关性,对Polymorph结果进行排序。
选择hydantoin-mol.xsd中的O=C-NH片段。在Functional Groups对话框中,确保选择Functional Groups作为Set。 单击“将选定的原子添加到集合Add selected atoms to set”按钮,然后单击3D Viewer中的任意位置以取消选择所选部分。乙内酰脲分子应如下所示: 接下来,将定义施主和受主位点。 在Functional Groups对话框中,将Set更改为受主Acceptors。在定义的官能团中选择氧原子,然后单击Add selected atoms to set按钮。 将Set更改为施主Donors,选择官能团中的氮原子,然后单击Add selected atoms to set按钮。关闭对话框。
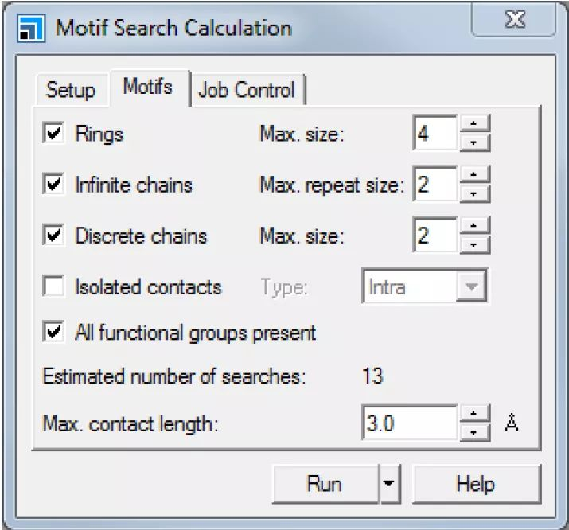
乙内酰脲分子中的官能团 4、利用CSD查询相关结构 在本节中,将在CSD中查询包含已定义官能团的结构,并提取施主和受主位点之间的连接信息。 从菜单栏中选择Modules | CCDC | Motif Search,打开Motif Search Calculation对话框。 在Setup选项卡中,将Task更改为CSD Search,确保勾选Return CSD hit structures复选框。 在Motifs选项卡中,勾选Rings、Infinite chains、Discrete chains和All functional groups present复选框。将Rings的Max. size设置为4,Infinite chains的Max. repeat size和Discrete chains的Max. size均设置为2。将Max. contact length设置为3.0Å。
在Job Control选项卡中,从Gateway location下拉列表里选择合适的服务器。单击Run按钮。 查询完成后,结果将自动从服务器下载并存储在名为hydantoin-mol Motif Search的文件夹中。匹配结构及其基序公式存储在数据表文件中。还创建了一个概要文档hydantoin-mol Summary.txt。在下一节中,当比较Polymorph预测的结构和CSD查询的结构时,将使用此概要文档作为评分任务的输入。
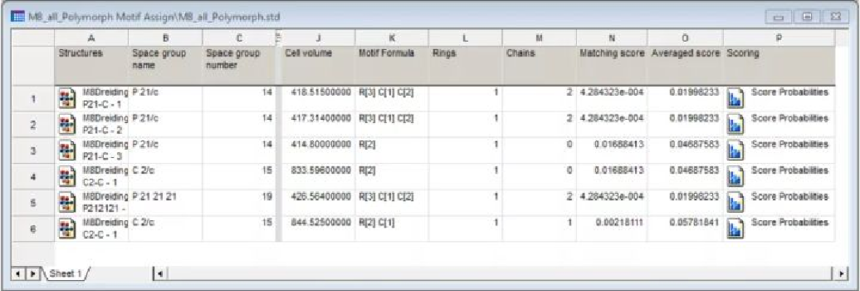
5、为Polymorph输出分配基序信息,并评分排名靠前的Polymorph结构 由于在本教程中未运行多晶型预测序列,因此需要从之前的预测序列中将结果导入到本工程中。如果想自行尝试预测堆积排列,建议利用DMol3模块的GGA-PBE泛函对乙内酰脲的几何构型进行优化,以确定ESP电荷,然后使用这些电荷和Dreiding力场运行Polymorph。有关如何运行多晶型预测序列的详细信息,请参阅Polymorph模块教程部分。 从菜单栏中选择File | Save Project,然后选择Window | Close All。 从菜单栏中选择File | Import...,打开Import Document对话框。导航至Examples/StudyTables,在文件类型下拉列表中选择所有文件All Files,然后选择M8_all_Polymorph.std。单击Open按钮。 接下来,将设置输入参数以对预测的堆积排列进行分配和评分。注意,要运行分配/评分,必须将包含预测结构的数据表文档打开,并且需要在界面中选择包含片段/接触点信息的文档。 在Motif Search Calculation对话框中,将Task更改为Assign Motifs,并从Scoring下拉列表中选择CSD。从Functional groups下拉列表中选择hydantoin-mol.xsd。勾选“使用现有CSD搜索Use existing CSD search”复选框,并从Summary document下拉列表中选择hydantoin-mol Summary.txt文件。 在Motifs选项卡上,确保参数设置与上一节中相同。单击Run按钮并关闭对话框。 计算完成后,将返回结果。主要结果文档是M8_all_Polymorph Motif Assign文件夹中名为M8_all_Polymorph.std的数据表。它列出了原始结构及其基序公式。前两个结构为相同的基序公式R[3] C[1] C[2],并且具有Polymorph计算得到的最低能量。第三和第四种结构的基序公式为R[2]。查看匹配得分Matching score和平均得分Averaged score,第三和第四个结构的基序明显更能代表先前已知的包含相同片段的结构。注意,第三种结构是实验上已知的结构。 注意:数据表中的值可能因CSD系统软件版本而有所差异。 双击基序公式为R[2]的第三和第四个结构的Structure单元格。 将打开一个新的3D Viewer,其中包含结构Polymorph预测得到的单位晶胞,分配的氢键基序以粉色虚线显示。 从CCDC Motif搜索计算中获得的评分值表明,第三和第四个结构是可以进一步研究的候选结构,不应被忽略,因为还有其他能量较低的结构。例如,可以使用Reflex模块模拟X射线粉末衍射图谱,并与实验图谱进行比较。
使用基序搜索Motif Search对前6个Polymorph预测结构进行评分的结果 |