|
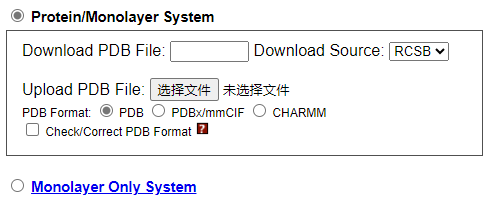
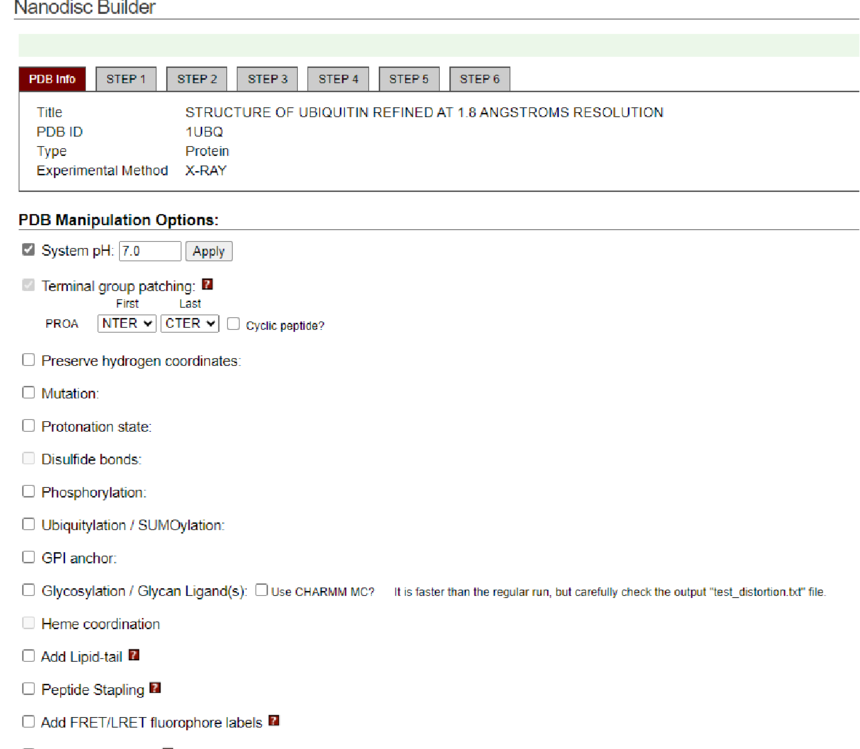
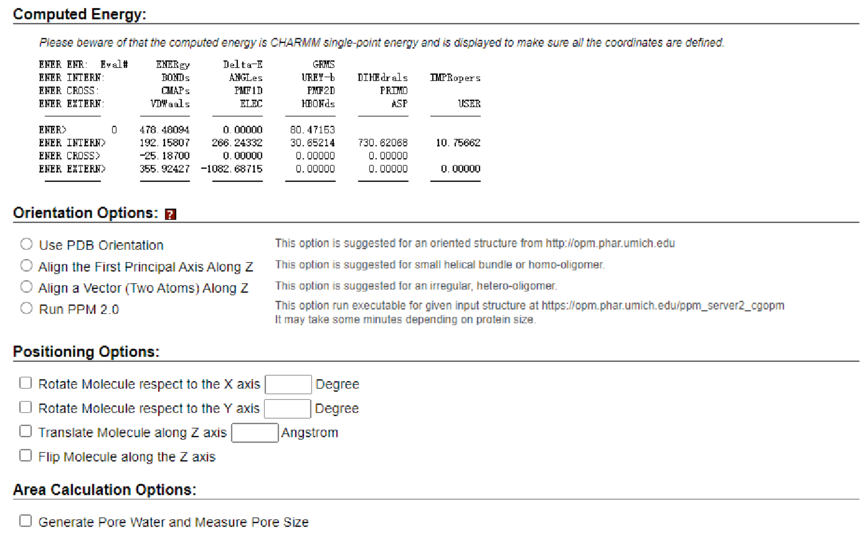
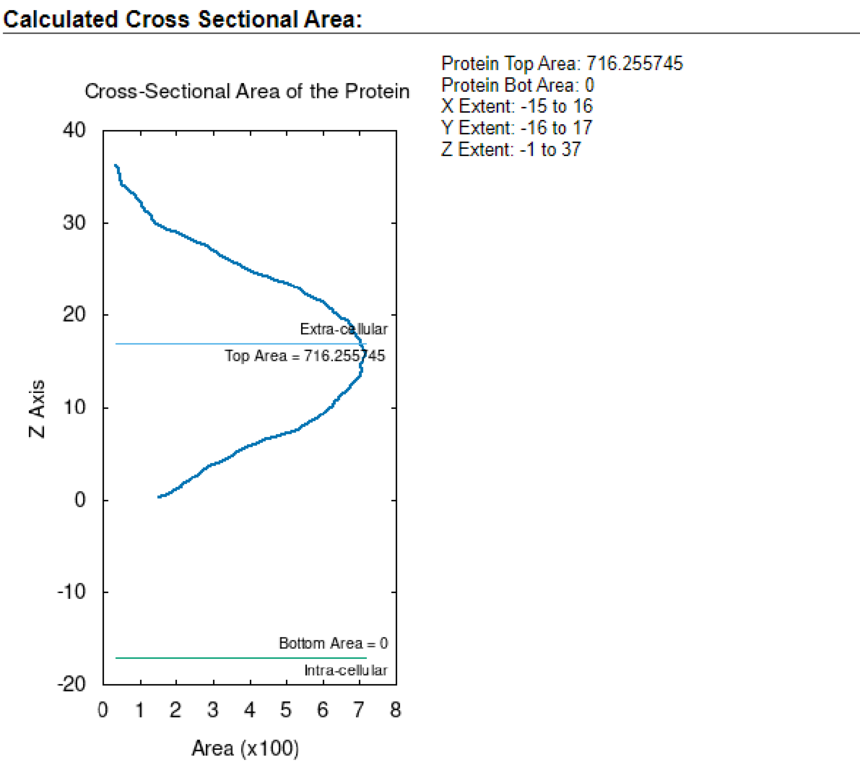
Monolayer BuilderMonolayer Builder帮助用户生成构建用于分子动力学模拟的蛋白质/单层(或仅单层)系统所需的一系列CHARMM输入。下面给出了每个步骤的简要描述。 在各种其他构建方案中,用户可以在步骤3中选择“插入”或“替换”方法。(用户可以在步骤3中选择其中之一,见下文)。 ■插入方式 将蛋白质插入具有大小与蛋白质大小相当的孔的预平衡脂质双分子层中(脂质双层的文库可在档案中获得) ■更换方式 蛋白质首先由脂质样球体包装,其位置随后用于从库中随机选择脂质分子(脂质分子库可在档案中获得) 单分子层系统的构建过程与双分子层系统相同,只是在步骤5中使用周期性边界条件将双分子层系统转换为单分子层系统(参见转换步骤)。 请注意
参考文献 S. Jo, T. Kim, V.G. Iyer, and W. Im (2008) CHARMM-GUI: A Web-based Graphical User Interface for CHARMM. J. Comput. Chem. 29:1859-1865 S. Jo, T. Kim, and W. Im (2007) Automated Builder and Database of Protein/Membrane Complexes for Molecular Dynamics Simulations. PLoS ONE 2(9):e880 S. Jo, J.B. Lim, J.B. Klauda, and W. Im (2009) CHARMM-GUI Membrane Builder for Mixed Bilayers and Its Application to Yeast Membranes. Biophys. J. 97:50-58 E.L. Wu, X. Cheng, S. Jo, H. Rui, K.C. Song, E.M. Dávila-Contreras, Y. Qi, J. Lee, V. Monje-Galvan, R.M. Venable, J.B. Klauda, and W. Im (2014) CHARMM-GUI Membrane Builder Toward Realistic Biological Membrane Simulations. J. Comput. Chem. 35:1997-2004 J. Lee, X. Cheng, J.M. Swails, M.S. Yeom, P.K. Eastman, J.A. Lemkul, S. Wei, J. Buckner, J.C. Jeong, Y. Qi, S. Jo, V.S. Pande, D.A. Case, C.L. Brooks III, A.D. MacKerell Jr, J.B. Klauda, and W. Im (2016) CHARMM-GUI Input Generator for NAMD, GROMACS, AMBER, OpenMM, and CHARMM/OpenMM Simulations using the CHARMM36 Additive Force Field. J. Chem. Theory Comput. 12:405-413 J. Lee, D.S. Patel, J. Ståhle, S-J. Park, N.R. Kern, S. Kim, J. Lee, X. Cheng, M.A. Valvano, O. Holst, Y. Knirel, Y. Qi, S. Jo, J.B. Klauda, G. Widmalm, and W. Im (2019) CHARMM-GUI Membrane Builder for Complex Biological Membrane Simulations with Glycolipids and Lipoglycans. J. Chem. Theory Comput. 15:775-786 J. Lee, M. Hitzenberger, M. Rieger, N.R. Kern, M. Zacharias, and W. Im (2020) CHARMM-GUI supports the Amber force fields. J. Chem. Phys. 153:035103 第1步
第2步
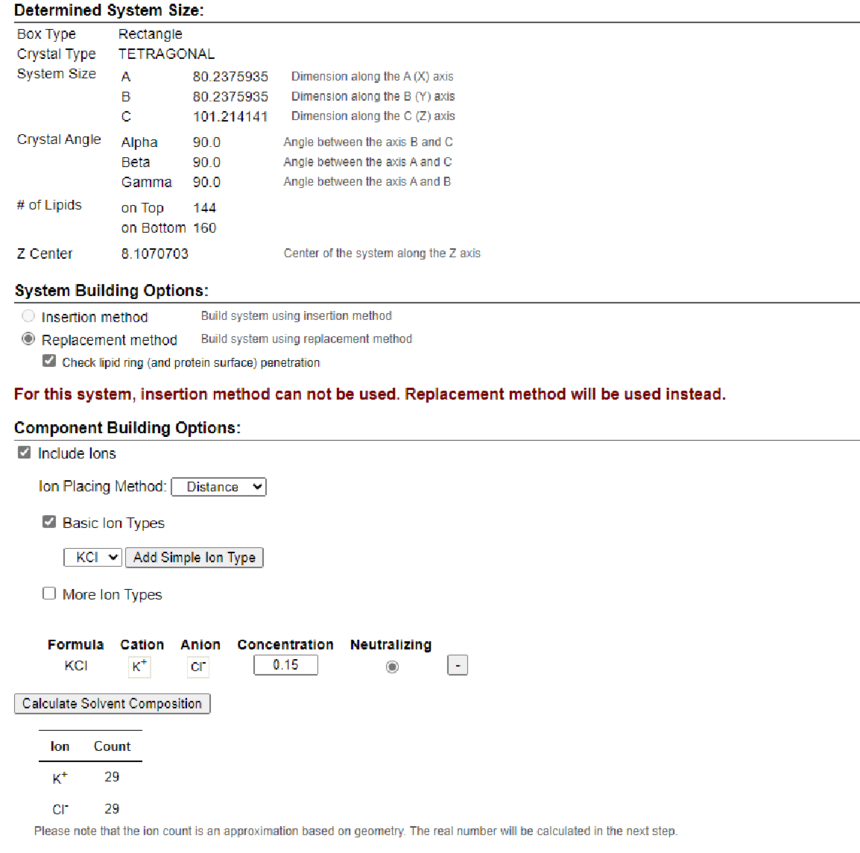
第3步
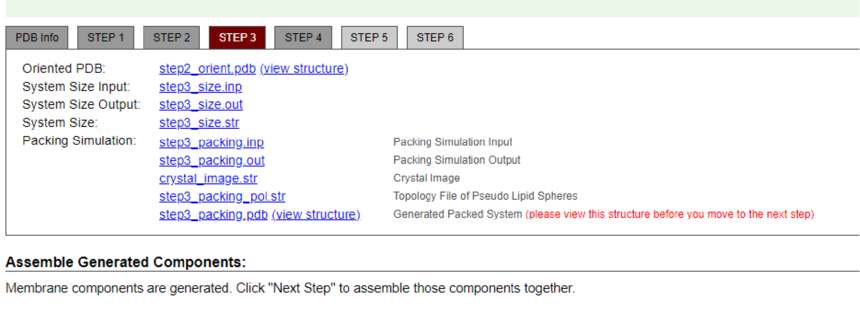
第4步
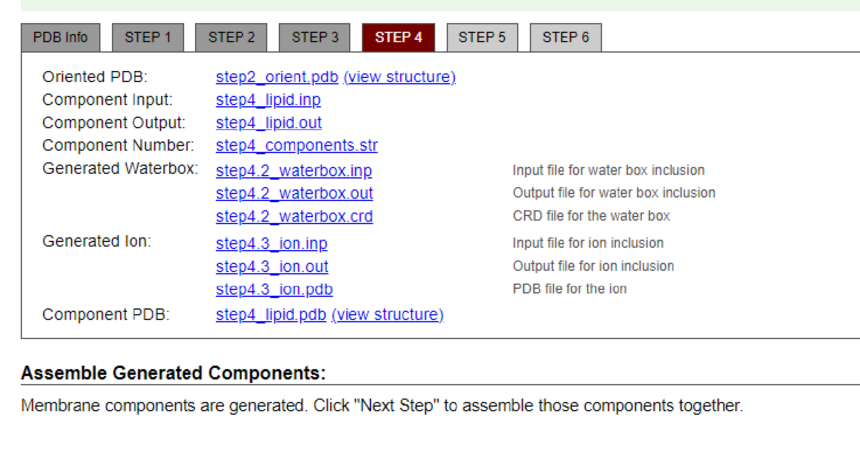
第5步
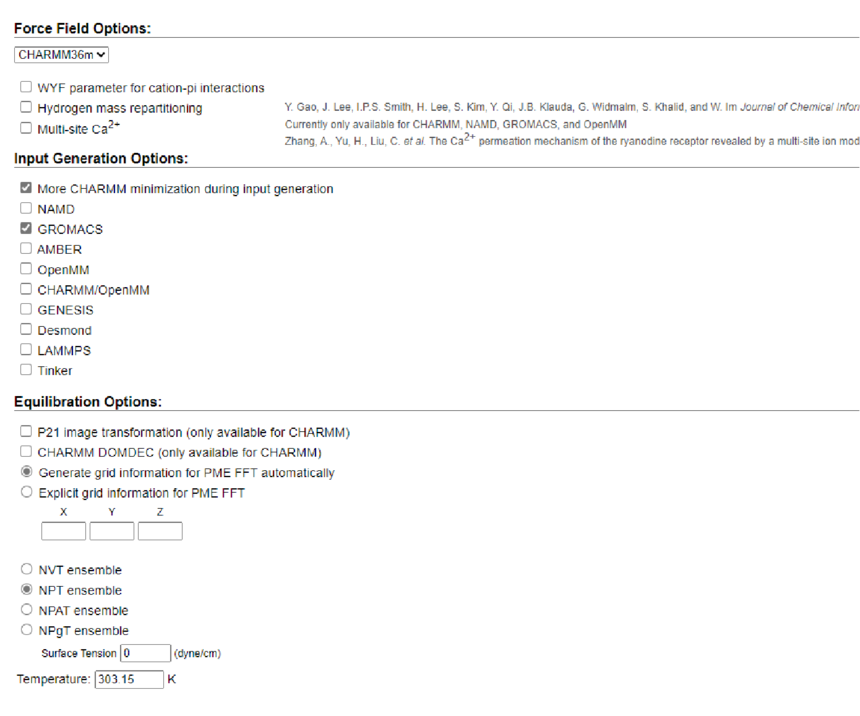
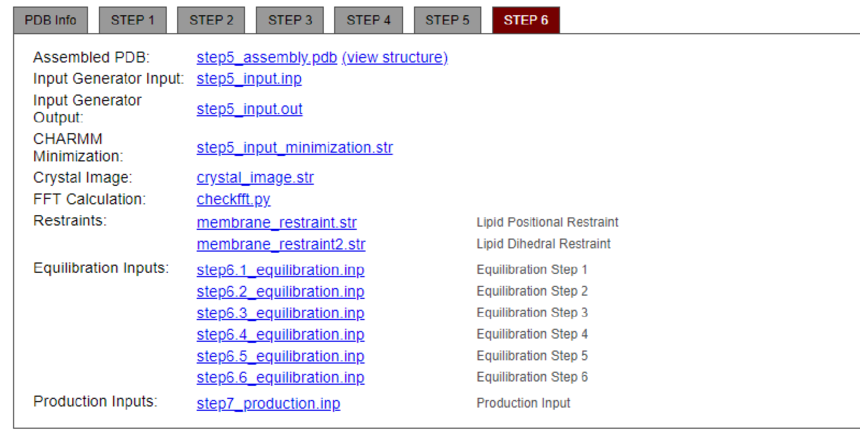
第6步
第7步
第8步
|