|
势能时间:2025-05-06 前面我们知道要获得所有原子的轨迹,需要知道原子的质量,初始位置,初始速度和依赖于位置的受力函数。原子的质量,初始速度和初始位置都是我们人为设定的值。不同研究对象的原子质量,初始速度和位置很容易获得。不同研究对象更本质的差别在于依赖于位置的受力函数,这个函数又被称为相互作用。相互作用的形式和参数决定了研究对象的性质和行为。在分子动力学中,这个相互作用通过原子之间的势能进行描述,因此又称为势能函数或势函数。而受力是势能对位置的负梯度,因为实践表明原子之间的受力都是保守力。举个例子,重力和重力势能。取竖直向上为正方向,则重力表达式为
重力势能的表达式是
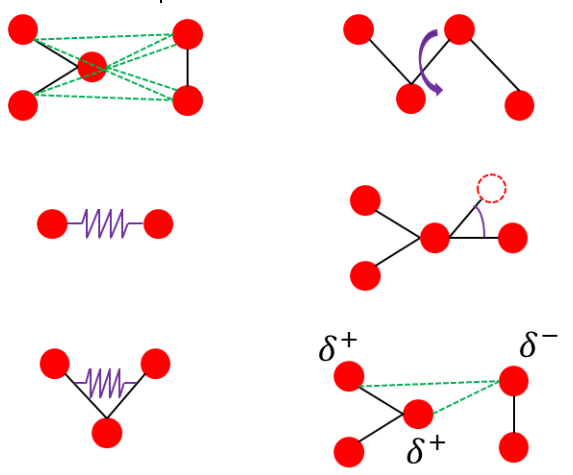
即重力势能对位置的负梯度就是重力。为什么要用势能,而不是受力来表达相互作用呢?这是因为在绝大多数模拟中采用的是笛卡尔坐标。势能是标量,而力是矢量,数学上处理起来更容易。在计算受力的时候,直接用势能对两个原子之间的相对位置进行求导,然后将这个力乘以方向余弦就能得到xyz三个方向上的受力。举例说明,在三维空中有两个原子
两个原子之间的距离
那么j原子对原子i的受力就为
这样不管是程序的简洁性还是计算效率都会很好。 所以要开展分子动力学计算,首先必须确定所有原子之间的势能函数,即势能关于两个原子之间相对位置的函数。确定函数分为两个部分:函数表达式和表达式中的常数值。对于一个对象,描述其相互作用的势能函数表达式和表达式中常数值的集合称为该对象的力场。通常一种力场可以用来描述一类对象。分子动力学模拟一般研究的对象可以分为:惰性气体,分子物质,金属晶体和非金属晶体。分子动力学模拟绝大多数情况用来研究固体和液体,即凝聚态。对于气体分子动力学模拟效率太低,有更好的办法进行研究。 简单液体 原子之间相互作用只具有范德瓦尔斯作用的物质。描述范德瓦尔斯作用最常用的表达式是Lennard-Jones12/6势能。通常用LJ12/6描述的物质也称为简单液体。
上图是LJ/126势函数的图像。对于该势函数有几个重要的点要说一下。势能的最低点的到零点之间的绝对值为 分子物质 原子之间有化学键连接的物质。比如常见的水,甲烷,乙醇,醋酸,氯化钠,聚乙烯,酯类,氨基酸,RNA,DNA等。分子物质是分子动力学模拟应用最广的对象之一。为了描述分子物质研究者开发出了一系列力场。值得说明的是力场是一个经验性的东西。研究者通过自己的物理直觉和反复试验,最终建立起适合某一类物质的力场。对于分子物质,相互作用包括两类:非键结势能和键结势能。非键结用来描述没有化学键连接原子之间的相互作用,键结势能用来描述有化学键连接原子之间的相互作用,如下图。
在一个体系中任意两个非键结的原子,如果其距离小于截断半径那么都要考虑它们之间的非键结相互作用。对于大多数力场非键结势能用LJ12/6描述。对于键结原子之间的相互作用则通过键结势函数进行描述。键结相互作用分为键能(bond),键角能(angle),二面角能(dihedral),离平面能(improper)。两个以化学键相连的两个原子都具有键能相互作用,当化学键偏离平衡长度的时候两个原子就会出现相互作用。两个化学键连接的相邻原子之间均具有键角能,当三个原子之间的角度偏离平衡角度的时候就会产生键角能。四个化学键连接的相邻原子之间会存在二面角能相互作用,当四个原子构成的二面角偏离平衡二面角的时候四个原子之间会出现二面角相互作用。四个相邻化学键连接的相邻原子之间有时还会存在离平面势能作用,当第四个原子偏离前三个原子所构成平面的平均夹角时,四个原子之间会存在离平面能相互作用。不同的分子会具有不同的键结势能。如氧气分子就只具有键能相互作用,水分子具有键能和键角相互作用,十二烷分子具有键能,键角能和二面角相互作用分子。元素构成较为复杂的分子,如RNA,就会同时具有四种键结相互作用。具体一个分子具有哪些键结能相互作用,由描述该分子的力场所定义。你只需要使用合适的软件在建模的时候会自动定义好各种键结信息和势能函数的参数。值得指出的是,在分子体系中原子一般都带有电荷,所以非键结原子之间会存在静电相互作用。在大多数力场中,原子的电荷都是部分电荷,也就是电荷一般都是零点几几几。这是由于在共价键中任何一个原子都并没有完全得到或失去一个电子,只是部分的得到或失去电子,因此在描述其电荷的时候都是零点几几几。少数强电解质会有整数电荷,比如氯化钠中的氯离子和钠离子,分别是-1和+1电荷。这里的电荷单位是一个电子或质子所带的电荷量,也就是单位电荷量。 为什么会采用多种键结势能函数来描述分子体系呢?这是因为键结的原子之间电子云的相互作用会对分子的构型进行限制,导致分子会具有特定的几何形状。分子动力学为描述这种情形就采用这样键能组合的形式。这是一种经验的方法。实践表明这是一种很好的办法。只要有合适的势函数形式和参数,采用的力场就能够准确描述分子的构型和之间的相互作用。目前研究者针对分子体系已经开发出多套力场。李继存老师对分子体系的力场有一篇文章做了很好的总结,现引述如下(具体网址见科学网—力场与拓扑之二:如何选择力场 - 李继存的博文 (sciencenet.cn)): 性能 模拟有机小分子热力学性质用Charmm generalized >= OPLS-AA >= GAFF,但实际上GAFF已经很好了。它们计算各种有机分子的密度、蒸发焓都很准确,但是介电常数、等温压缩系数计算得都一般。GAFF对于带有硝基的分子不好。OPLS和GAFF对于苯甲醛、甲酸,以及有两个及以上Br或Cl相距较近的情况都不好。 对蛋白质构象的模拟: ff99SB+ildn+nmr > CHARMM27 >OPLS>>f99。 Berger:专门用于磷脂的力场 PFF=Polarizable Force Field VAMM=Virtual atom molecular mechanics 材料力场 cvff(consistent valence forcefield):参数用于有机分子、蛋白质模拟,函数形式略复杂。cvff_aug是对其扩展,可以用于研究硅酸盐、铝硅酸盐、磷酸盐、泥土 CFF(consistent family of forcefield):包括CFF91和CFF95。适用面很广,涵盖有机无机小分子、聚合物、多糖和生物大分子,还支持金属。函数形式挺复杂。参数由从头算获得,非键参数从CVFF弄来,不适合凝聚相模拟。 pcff:基于CFF91,适用范围做了扩展,主要用于聚合物和有机材料,也能用于无机材料,还有糖、核酸、脂的参数。 COMPASS=Condensed-phase Optimized Molecular Potentials for Atomistic Simulation Studies:在pcff基础上改进的新版本,同样由从头算获得参数,在凝聚相模拟方面大有改善。适用于有机和一些无机分子、高分子,常用于材料领域的各种性质计算,不支持生物分子。模拟超临界水不错。能够适应很宽范围的压强和温度。MS中COMPASS(即2.8)>COMPASS2.7>COMPASS2.6。在MaterialStudio中御用,参数是加密不公开的,虽然lammps也能用,但是参数不全。 普适力场 Dreiding:普适型力场,但支持的元素有限,并非涵盖整个周期表。可以用于有机、生物、主族无机分子。结构、结合能的计算结果精度一般。没有指定计算电荷的方法,建议用拟合静电势电荷,原文用Gasteiger勉强应付。 UFF=Universal Force Field:涵盖整个周期表的普适型力场。比Dreiding更好。函数不复杂。UFF计算结构、结合能的计算结果精度一般,主要适合找不到适合的力场时凑合用。默认用QEq电荷。缺少纯金属单质的参数,都是离子的。 反应力场 REBO:反应力场,主要用于固体,无非键参数。后来添加了C、H的LJ参数的叫做AI-REBO力场。 ReaxFF=Reactive force field:ADF、MS、lammps支持,作者本人也有相应的程序但不公开。这是反应力场,引入键级概念,可以研究模拟过程中的设计拓扑结构改变的化学反应,挺流行。对于结构新鲜的体系可能差一些。用于爆炸、燃烧过程的反应貌似不错。支持所有主族和部分过渡金属元素。电荷由EEM获得。参数很多很复杂难找全。形式复杂,步长需要比较小,比如0.1~0.5fs。计算速度慢,貌似介于半经验与一般分子力场耗时之间,只适合最多用于几千个分子,再多的话就得和普通力场联用(类似QM/MM)。 专业小分子力场 MMX:早期的有机小分子力场。 MM2/MM3/4=Molecular Mechanics versions 2/3/4:用于小分子。函数复杂。计算小分子结构能量好,适合构象搜索、计算频率、获得最稳定结构。由于VDW参数不好,凝聚相问题差。MM3是这一类中最流行的。MM3也有蛋白质参数,叫MM3PRO。 MMFF=Merck Molecular Force Field:各方面类似于MM2/3,但比之更好。在CFF之后才发展的,也是从头算计算参数,但提升至MP2级别。参数比较细而且广泛。凝聚相问题仍不好。 大分子力场 CHARMM=Chemistry at HARvard Macromolecular Mechanics,函数形式简单,包含以下版本: CHARMM19:联合原子力场,也适用于蛋白质。 CHARMM22:全原子力场,适用于蛋白质。 CHARMM22/CMAP:适用于隐势GBSW。名字编号是最初使用这种力场的CHARMM版本得名的。 CHARMM27:适用于核酸和脂(膜)。与22结合可以混合使用模拟蛋白+核酸体系。应当用TIP3P,是在这种水模型下拟合的。 CHARMM General Force Field(CGenFF):用于药物类小分子,也可视为通用有机小分子力场。可结合其它CHARMM全原子力场使用。 CHARmm:CHARMM的商业版本,归Accelrys。 AMBER=Assisted Model Building with Energy Refinement:适合蛋白和核酸的凝聚相模拟,有机小分子支持得少。函数形式简单。包含以下版本: ff12力场(parm10.dat+frcmod.ff12SB):与ff10的区别是对蛋白的骨架和侧链扭转项参数做了进一步修正以更吻合实验。核酸的参数没变。这是目前最好的amber力场。 ff10力场(parm10.dat):对ff99的各种参数补丁的集合,相当于parm99.dat+frcmod.ff03+bsc0+chi.OL3+新的离子参数+原子和残基名的修改以顺应PDB format version 3。蛋白的参数和ff99SB相同。 ff99SBildn(frcmod.ff99SBildn):对ff99SB的异亮氨酸、亮氨酸、天冬氨酸、天冬酰胺的侧链参数改进的补丁。 ff99SBnmr(frcmod.ff99SBnmr):基于NMR数据对ff99SB的骨架参数进行修正的补丁。 ff03.r1力场(parm99.dat+frcmod.ff03):ff99力场的修改版。获取电荷时通过连续介电模型表现溶剂可极化效应,修改了蛋白phi、psi骨架参数,减少了对螺旋构象的偏爱。核酸参数相对于ff99没变。ff03.r1与amber9中的ff03略有不同,那时仍用的是ff94的方法得来的碳、氮端基原子电荷,如果仍想用那时代的ff03就调用oldff/leaprc.ff03. ff03ua力场(parm99.dat+frcmod.ff03+frcmod.ff03ua):ff03力场的united-atom版本,侧链的氢原子被united了,骨架上的氢原子和芳香环上的氢原子仍被保留。由于骨架还是全原子故骨架势参数没变,侧链上的参数因用了united故重新拟合。核酸参数完全没变,且还是全原子。 ff02力场(parm99.dat+frcmod.ff02pol.r1):ff99力场的可极化版,给原子上增加了可极化的偶极子。frcmod.ff02pol.r1是对原ff02的扭转参数的修正。 ff02EP力场(parm99EP.dat+frcmod.ff02pol.r1):ff02力场基础上给诸如氧、氮、硫原子增加了偏离原子中心的点电荷以表现孤对电子效应。据称比ff02稍好点。 ff99力场(parm99.dat):大部分参数来自ff94力场,修改了许多扭转角的参数。甘氨酸的骨架参数有问题,螺旋和延展构象的平衡性不对。而对于DNA,ff99长时间模拟中亚稳态占统治地位,即alpha和gamma二面角倾向于分别为gauche+和trans状态。虽然在RNA中也有这问题,但不严重。ff99的这些毛病在ff94里也有。 ff99SB力场(parm99.dat+frcmod.ff99SB):对ff99的蛋白二面角参数进行修正,二级结构间分布的比例得到了改善,也解决了甘氨酸骨架参数问题。 bsc0(frcmod.parmbsc0):解决上述ff99在核酸模拟问题上的补丁,同时还改进了RNA的糖苷的gamma二面角扭转势。可参考http://mmb.pcb.ub.es/PARMBSC0。 ff99SB+bsc0力场:把bsc0补丁用到ff99SB上,相对于ff99同时增进对蛋白和核酸的效果。这个组合使gamma二面角过分偏离了trans型。如果初始结构有很多gamma角为trans的情况,还是用ff99比较好。 ff99SBildn(frcmod.ff99SBildn):在ff99SB基础上修改氨基酸侧链参数的补丁。 ff99SBnmr(frcmod.ff99SBnmr):在ff99SB基础上修改骨架扭转项参数以更符合NMR数据的补丁。 ff98力场(parm98.dat):对ff94改进了糖苷的扭转角参数。 ff96力场(parm96.dat):与ff94扭转角不同,算出来的能量更接近量化结果。来自Beachy et al,由于构象有明显偏向beta等问题,使用不广泛。 ff94力场(parm94.dat):来自Cornell, Kollman et al。适合溶剂环境。电荷由RESP HF/6-31G*获得。 ff86力场(parm91X.dat):将ff84扩展为全原子力场。和ff84一样对氢键也是用Lennard-Jones 10-12势,故如果想在sander里用ff84/86,得重新带着-DHAS_10_12选项编译。之所以相应的文件叫parm91X是因为对原始ff86做了一些修正。(parm91X.dat是parm91.dat的补完版,加入了一些非键项,但非键项比如Mg、I等的参数都没调好,只是近似。) ff84(parm91X.ua.dat):最早的AMBER力场,用于模拟核酸和蛋白质的联合原子力场。不推荐使用,但在真空或者距离依赖的介电常数下模拟还有用。 parmAM1和parmPM3力场(parmAM1.dat/parmPM3.dat):用这个参数对蛋白质优化可以得出与AM1/PM3相同的优化结果。如今已没什么价值。 GAFF力场(gaff.dat)=Generation Amber Force Field:普适型有机小分子力场,函数形式和AMBER力场相同,与AMBER力场完全兼容。 GLYCAM-06力场(GLYCAM_06g.dat):对以前GLYCAM力场做了改进,并且纳入了一小部分脂类的参数。 GLYCAM-04EP力场(GLYCAM04EP.dat):将GLYCAM04扩展到可用于TIP5P模型下的模拟。给氧加上非原子中心点电荷表现孤对电子效应。 GLYCAM-04力场(GLYCAM04.dat)=glycans and glycoconjugates in AMBER:专用于糖的模拟,和AMBER完全兼容,可一起用于糖蛋白的模拟。官网:http://glycam.ccrc.uga.edu/ccrc/index.jsp lipid11:与Amber力场完全兼容的模拟磷脂和胆固醇的力场。 AMOEBA:可极化力场,能模拟水、蛋白、单价离子、有机分子。速度比起一般的固定点电荷力场慢8倍。 GROMOS=Groningen Molecular Simulation:适合烷烃、蛋白、核酸凝聚相的模拟,函数形式简单。A前边的数字代表此力场所含原子类型数目,后面的是版本号。 G43B1:适用于真空下的模拟,现在基本不用。 G43A1:是最早的GROMOS96力场,是联合原子力场(极化的氢才表达出来),用于凝聚相,后面那些都是对这个的小幅改进 G43A2:是改进了烷烃中原子类型的二面角参数 G45A3:是改了烷烃的VDW参数以更好地符合实验数据(气化焓、可压缩率、压强、水合热等) G45A4:是为了改进核酸的模拟,增加了二面角参数、原子类型、改动了原子电荷 G53A5和G53A6:调整了参数,分别使生物分子(蛋白、DNA、糖、脂)在环己烷和水中的溶解自由焓贴近实验值。但是蛋白模拟时的结构比起G45A4却有了缺陷。 G54A7/B7:修改了psi/phi的扭转角参数以改正模拟蛋白质时对螺旋的稳定性问题,加入了一个-CH3的原子类型,修改了Na+和Cl-以符合其水合能,加入了一个和手性改变相关的improper项。模拟蛋白质的稳定性比G53A6更好了。G54B7是用于真空模拟,是在G53B6基础上做了类似G54A7对G53A6的改进得到的。 OPLS=Optimized potentials for liquid simulations:Jorgensen搞的。适合有机小分子、蛋白凝聚相的模拟。函数形式简单。最初OPLS-UA是联合原子力场,现在一般都用OPLS-AA全原子力场。 MARTINI:流行的粗粒化力场,四个原子(氢也算)凑一个原子,可以做磷脂、蛋白质、聚合物、糖、胆固醇。在gromacs里可以用。一般25~40fs一步,不能考虑二级结构的变化,必须事先指定好。http://md.chem.rug.nl/~marrink/coarsegrain.html ENCAD=Energy Calculation and Dynamics:不流行。专门模拟溶液下蛋白和核酸,侧重于模拟过程中的能量保守性。 Shinoda2007=粗理化力场,专用于模拟水与表面活性剂体系,平均三个重原子折合一个bead。 水模型 不一定水模型是力场御用的就一定比用其它的水好。对于非极化的水,TIP4P-EW是最好的。对于可极化的水,AOMEBA是最好的。 TIP3P:amber、gaff御用的。 TIP4P:OPLS御用的。 TIP4P-EW:在TIP4P基础上专为ewald计算优化,对水的凝聚相各种热力学、结构性质表现得比其它水模型都好。 SPC:GROMOS力场御用的。 SPCE:对纯水体系比SPC、TIP3P都好。但是有人说这个在纯水下表现好,在混合体系疏水性略强。 shell水模型:在gmx中直接支持。就是给普通的水中间加了个振子表现可极化效应。虽然形式是所有可极化水模型里几乎最简单的,耗时少,但是步长必须设得很短,0.2fs。 AMOEBA水模型=Atomic Multipole Optimized Energetics for Biomolecular Application:主要在Tinker中使用,amber也支持。包括比较复杂的可极化水模型,是三点模型,在原子中心上除了单极矩(原子电荷)外还用偶极矩、四极矩来精确描述电荷分布,并且给予原子由拟合实验值得到的原子极化率,在外场下产生诱导偶极矩以使电荷分布能响应实际环境,氢的范德华作用也考虑进去。不是一般的LJ势。这个水是柔韧的,因此不要用约束算法固定住它的结构。这个水模型管在纯水液相状况下比TIP5P没什么改进还多花时间,但普适性比起其它模型都好得多,气相、溶液相都适用,而不是像固定点电荷水模型只在特定环境下才好。对结构、热力学数据、结合能重现得都好。 金属 磷脂膜模拟的力场 Gromos96:rtp本身自带了DPPC参数,结果不好。 CHARMM27及改进版CHARMM36c:专门且常用的膜力场。 Glycam06:支持了少数磷脂分子,非主流。 GAFF:GAFF力场没有膜的参数,直接用在膜模拟效果不好。 Lipid11(2012):Skjevik提出的膜力场,作为amber系列力场的扩展,参数来自GAFF,几种头部(PC,PE,PS,PH,P2,PGR,PGS,PI)和几种尾部可以自由搭配(模块化)组成磷脂,还支持胆固醇,完全兼容amber力场,leap已支持。非主流。Dickson(2012)的GAFFlipid力场只是一个阶段性的膜力场,将会被融合进Lipid11。 Berger(1997):联合原子膜力场。成键参数基于GROMOS87,LJ参数基于OPLS-UA,适合搭配Gromos87,很常用也很好,几乎是唯一致命的问题在于不直接兼容Gromos96,若搭配OPLSAA需要很留神。虽然也有一些人结合Gromos96来模拟膜蛋白,但终究比较古怪,需谨慎。原文只给出了DPPC的参数,后来又有人基于此弄了其它磷脂的。Berger本身没直接提供参数和拓扑文件,Peter Tieleman基于Berger的参数制作了DPC、POPC、DPPC、DMPC、DLPC、DOPC、PLPC、POPE的itp文件,都需要lipid.itp中的参数,可以在这里下载:http://wcm.ucalgary.ca/tieleman/downloads G43A1-S3 (2006):Chiu弄的兼容Gromos43A1的膜力场。支持PC/PE/sphingomyelin和cholesterol。此力场的POPC不建议使用。 Kukol(2009):完全兼容Gromos96 G53A6的膜力场,烷烃链是联合原子,结果很好,和Berger相仿佛,弥补了它不支持Gromos96的遗憾。拓扑文件从原文的补充材料里得到。包含DPPC、DMPC、POPG、POPC、DMPC的参数。此力场的POPC不建议使用。 DAVID POGER(2010):完全兼容gromos96 G53A6的膜力场。JCC的文章中只提出了DPPC的参数,JCTC的文章中还提出了DLPC、DMPC、DOPC、POPC的参数。网址和gmx的拓扑文件:http://compbio.chemistry.uq.edu.au/~david/research/lipids.htm Stockholm lipids (Slipids) (2012):Jambeck弄的全原子膜力场。兼容amber。支持DPPC、DLPC、DMPC、POPC、DOPC、SOPC、POPE、DOPE、sphingomylin、PG和PS头部集团、胆固醇。gromacs的拓扑文件和预平衡的结构从这里下:http://people.su.se/~jjm/Stockholm_Lipids/Downloads.html MARTINI:粗粒化。网址和gmx的拓扑文件:http://md.chem.rug.nl/cgmartini/index.php/downloads Lipidbook汇总了各种膜力场的参数:http://lipidbook.bioch.ox.ac.uk ATB带了几十种兼容gromos96的膜参数(可能对应的gromos96版本不同),ATB也能自动生成新的磷脂的参数。ATB网址和gmx的拓扑文件:http://compbio.biosci.uq.edu.au/atb/ 测试总结(http://md.chem.rug.nl/cgmartini/index.php/blog/269-jungle2):slipid最好,CHARMM36其次,berger很好,Kukol和poger说得过去,后者貌似相对略好。磷脂平均表面积、平均体积、头部集团距离都很容易算准,所有膜力场都没问题,但扩散能力、等热表面可压缩率大多膜力场难以算准。值得一提的是,一种膜力场当中,某种磷脂算得好绝不代表另外磷脂就算得好。 金属晶体和非金属晶体 这两类物质具有特殊的结构,研究者为它们开发了额外的势函数,称为多体势函数,来描述原子之间的作用。所谓多体就是说两个原子之间的相互作用不仅取决于这两个原子还与其周围的原子有关系。针对某种物质研究者已经开发出相应的力场参数,只需要搜索一般就能找到对应的势函数参数或文件。我对这些势函数不太了解,就不在这里赘述了。 以上就是关于势能和势函数的内容。需要指出的是为了提高模拟精度,研究者还针对开发出了针对某一种物质的势函数。当你要模拟这些物质时,直接查找原始文件中的参数就行。关于力场的选择,当然是选针对要研究体系的力场。但是对于LAMMPS来说建模是个很麻烦的事。如果你能找到一种力场描述的体系,并且成功建模,那你就偷着乐吧。不同力场之间的差别并不是那么明显。事实上,分子动力学模拟最大的价值就在于给出研究对象的一般性规律和背后的机理,而不是计算出某些精确的数值。而大多数力场能够捕捉研究对象的本质特征,这就基本上能够保证这个力场能够再现研究对象的本质规律,这对于我们来说就已经足够了。当然,力场的开发一直在发展中以越来越精确的描述研究对象。比如几年来发展出来的机器学习力场就能够以量子的精度开展分子动力学模拟。这是分子动力学模拟方法中的一大进展。当你要验证一个力场的时候,计算研究对象你所关心的物理量的数值和实验或者文献对比,如果接近,力场就能用。 感谢鲍路瑶老师的分享,内容来自于鲍老师分享出来的资料 如有需要添加微信:lmp_zhushou 进入微信群,帮助他人,共建社区 获取完整版lammps讲义可以加微信lmp_zhushou或加入QQ群994359511 上一篇温度、压强下一篇gmx xpm2ps |


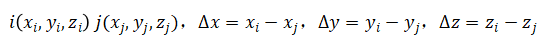
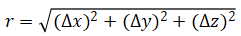
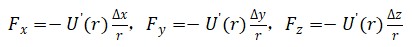
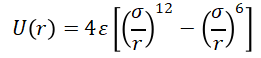
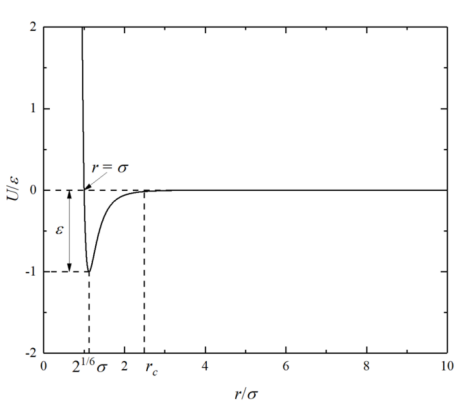
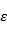 ,称为势能函数的特征能量,势能的零点位于处
,称为势能函数的特征能量,势能的零点位于处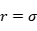 ,
,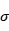 称为势能函数的特征长度。势能函数的最低点位于
称为势能函数的特征长度。势能函数的最低点位于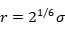 处,在该点处势能对位置的导数是零,所以该点处受力也为零。该点也称为平衡位置,我们可以看到在平衡位置出势能是负的。所以当体系处于平衡状态时,原子之间的相对距离大概也在平衡位置附近,所以势能为负的。这也就是为什么大多数情况分子动力学算出来的势能都是负的。在
处,在该点处势能对位置的导数是零,所以该点处受力也为零。该点也称为平衡位置,我们可以看到在平衡位置出势能是负的。所以当体系处于平衡状态时,原子之间的相对距离大概也在平衡位置附近,所以势能为负的。这也就是为什么大多数情况分子动力学算出来的势能都是负的。在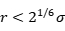 ,两个原子之间表现为相互排斥,在
,两个原子之间表现为相互排斥,在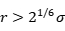 ,两个原子之间表现为相互吸引。从势函数的图像上可以看出在
,两个原子之间表现为相互吸引。从势函数的图像上可以看出在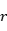 较大的时候,势能趋于零,其导数也趋于零。也就是当两个原子距离很远的时候,原子之间的相互作用可以忽略不计。因此在实际计算的时候都会使用截断的LJ/126。截断距离成为截断半径,其值大概在
较大的时候,势能趋于零,其导数也趋于零。也就是当两个原子距离很远的时候,原子之间的相互作用可以忽略不计。因此在实际计算的时候都会使用截断的LJ/126。截断距离成为截断半径,其值大概在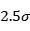 。就像重力势能一样,势能的计算需要一个参考位置。LJ/126的参考位置选择在了无限远处。类似LJ/126势能以及其他拓展的LJ势能,都称为对势,其含义是两个原子之间的相互作用只由这一对原子决定,与其它相邻原子没有关系。
。就像重力势能一样,势能的计算需要一个参考位置。LJ/126的参考位置选择在了无限远处。类似LJ/126势能以及其他拓展的LJ势能,都称为对势,其含义是两个原子之间的相互作用只由这一对原子决定,与其它相邻原子没有关系。