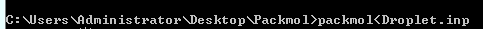|
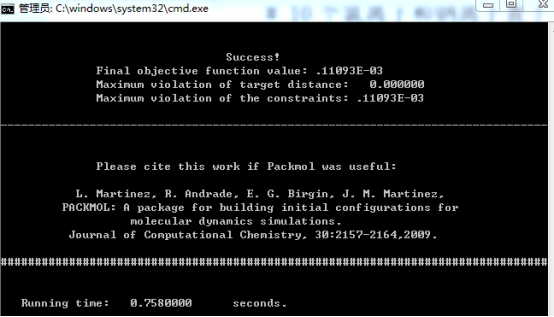
润湿-MS+packmol建模时间:2025-05-06 所需软件 目前市面上充斥着各种建模、模拟、后处理和绘图软件,我们不可能面面俱到,每个软件都会。所以只挑选了几个上手简单,又可以完成相对比较复杂的功能的软件。 建模工具包括Packmol,Material Studio和Atomsk。Packmol(http://leandro.iqm.unicamp.br/m3g/packmol/home.shtml)使用方法非常简单,只需少量脚本就可以建立比较复杂的模型,如下图所示。我们将使用Packmol建立所需形状的各类液滴,比如水滴、离子液滴、溶液液滴等。
Atomsk(https://atomsk.univ-lille.fr/index.php)用于构建具有复杂结构的基底。类似于Packmol,Atomsk同样具有容易上手,脚本简单,功能强大的特点,特别对于各种晶体模型(无论2D或者3D)的建立很方便。在润湿模拟,基底通常具有很多微观结构,使用Atomsk无疑可以达到事半功倍的效果。
Material Studio用于一些简单的建模、模型组装以及加力场,也可以用于可视化出图。 模拟主要靠LAMMPS全家桶,包括lammps主程序以及一些附件工具,如msi2lmp等。
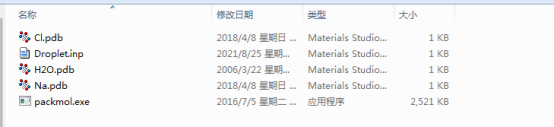
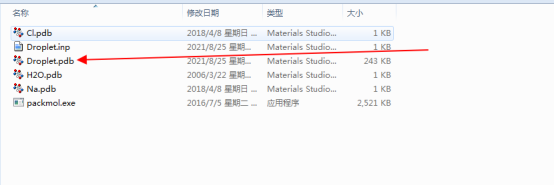
可视化和后处理工具主要包括OVITO和VMD,我们还需要会一些简单的脚本和小程序。 使用Packmol建立液滴模型 使用Packmol建立液滴模型需要以下几个文件:Packmol执行程序、分子模板文件和Packmol脚本文件。 Packmol源代码和执行文件可以在官网获取,我们也在QQ群(670911765)里上传了,需要的同学在群文件中自行下载。 http://www.ime.unicamp.br/~martinez/packmol/userguide.shtml Packmol语法非常简单,堪称一看就会,一做就对,就没有专门讲解的必要了,如果同学们有需求可以专门写一期。 现在准备分子模板,分子模板可以认为是我们需要建立模型的最小单位,比如水滴的分子模板就是水分子。如果液滴中含有不同分子、或者离子,则分子模板也需要分开准备。下面以纯水液滴和氯化钠溶液液滴为例。 水分子模板 HEADER water COMPND SOURCE HETATM 1 H HOH 1 9.626 6.787 12.673 HETATM 2 H HOH 1 9.626 8.420 12.673 HETATM 3 O HOH 1 10.203 7.604 12.673 CONECT 1 3 CONECT 2 3 CONECT 3 1 2 END 水分子需要给出原子序号、类型和相对位置(HETATM开头的行),以及连接情况(CONECT开头的行),其余行应该每个分子模板都是一样的。 氯离子模板 REMARK Materials Studio PDB file REMARK Created: Sun Apr 08 01:52:21 中国标准时间 2018 ATOM 1 Cl MOL 2 -0.457 1.541 18.284 1.00 0.00 Cl1- TER 2 钠离子模板 REMARK Materials Studio PDB file REMARK Created: Sun Apr 08 01:52:05 中国标准时间 2018 ATOM 1 Na MOL 2 0.653 1.541 18.284 1.00 0.00 Na1+ TER 2 氯离子模板和钠离子模板仅需给出离子的位置就行(ATOM开头的行),可以随意修改。细心的同学可以看出水分子模板和氯离子、钠离子的模板格式有些不同,那么笔者是怎么知道该怎么写呢?事实上,我也不知道,其实也不用关心,因为这个模板是从Materials Studio里面导出出来的。只需在Materials Studio建立一个水分子、氯离子或者钠离子在导出pdb格式文件即可,需要注意的事这个时候并不需要建立box。所以读者可以自行建立pdb文件,也可以采用本文给出的模板,将模板内容复制进入文档,并分别命名为H2O.pdb、Cl.pdb和Na.pdb即可。 Packmol脚本文件 下面开始准备Packmol脚本文件,本文以球形液滴为例。Packmol脚本文件的逻辑非常简单,即:读入分子模板==>把指定数量的分子放入指定区域==>写出模型文件。以下是Packmol脚本文件示例: # #表示注释 tolerance 2.0 #tolerance表示距离约束容差,可以理解为原子间最小距离不低于该值,对于水分子和氯离子钠离子来说2Å是个不错的值 filetype pdb #filetype表示原子类型,这里选择pdb格式 output Droplet.pdb #output表示输出文件,droplet.pdb为输出文件名 # 1000个水分子置于半径为20球内部 # 球心位于原点 structure H2O.pdb #以H2O.pdb为模板建立structure number 1000 #水分子数量为1000 inside sphere 0.0 0.0 0.0 20.0 #inside sphere表示在球内部,0.0 0.0 0.0 为球心坐标,20.0为半径 end structure #停止使用该模板建模 # 10个氯离子和钠离子置于半径为20球内部 # 球心位于原点 structure Cl.pdb number 10 inside sphere 0.0 0.0 0.0 20.0 end structure structure Na.pdb number 10 inside sphere 0.0 0.0 0.0 20.0 end structure 使用packmol和MS建模 将该脚本命名为Droplet.inp,将Packmol执行程序、分子模板文件和Packmol脚本文件放进同一个文件夹,如下图:
打开命令窗口,使用以下命令即可完成建模。 Packmol<Droplet.inp
文件夹中已经输出模型文件Droplet.pdb
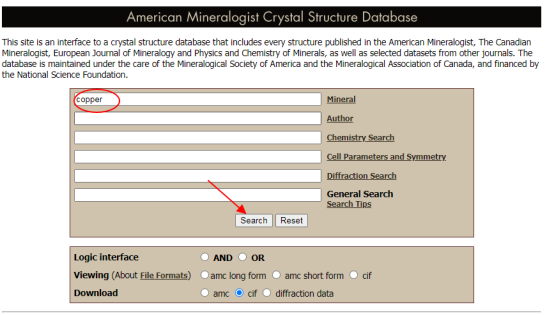
用Materials Studio打开即可。 使用MS建立基底+基底液滴合并 本期是润湿系列教程的第三期,主要讲解使用MS建立基底,并与上期使用Packmol建立的液滴合并,导出之后即可进行下一步操作,即模拟。 MS建立基底 在实际的模拟,基底常常会有不同的结构,使用atomsk可以轻易得到,我们在这里不讲解关于atomsk的操作。假设基底具有为原子级光滑的表面,首先需要得到基底的晶胞坐标,也可以直接下载晶胞的结构文件,然后在MS中进行扩胞,得到需要的尺寸即可,在此我们采用后一种方法。 假设润湿过程为:纳米水液滴在铜基底上润湿,所以我们先得到铜的cif文件,可从以下网址获取: http://rruff.geo.arizona.edu/AMS/amcsd.php 在Mineral一栏中输入:copper,然后点击search即可
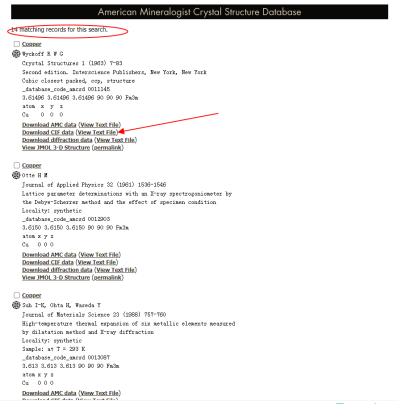
得到结果如下图所示,有14个搜索结果,都大同小异,我们选择第一个结果,点击下载CIF data即可。
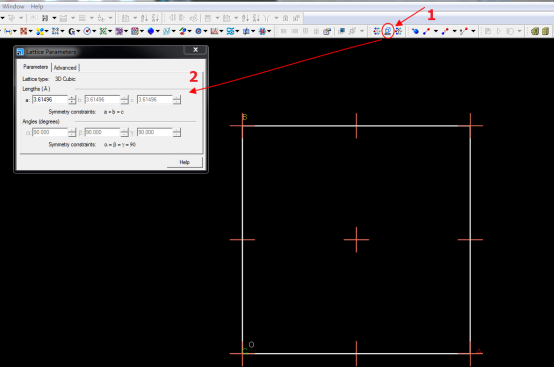
使用MS打开下载的cif文件,点击如图所示的小立方体,就可知晶胞的参数,三个长度数据和三个角度数据,此案例中a=b=c,三个角度均为90度。
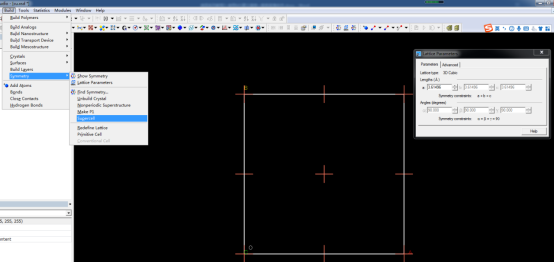
我们假设润湿模拟中基底的a、b方向长度为50*晶胞棱长,c方向为3*晶胞棱长,所以需要对该模型进行扩胞,即建立supercell。路径为:Build>Symmetry>supercell。
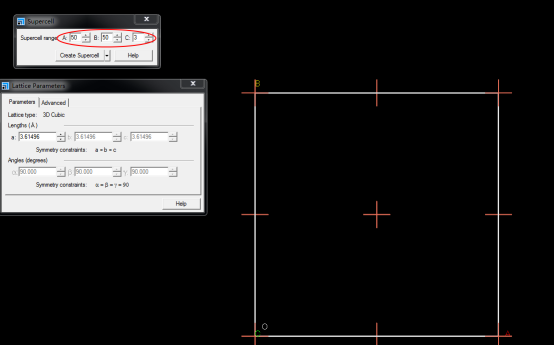
点击之后如下图所示,输入所需的倍数即可,这里为50*50*3。
得到模型,如下:
换个显示角度:
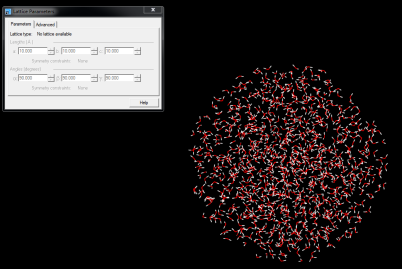
建立液滴模型 建立球状液滴,建立方法在教程二中已有说明,为了使液滴和基底可以顺利合并,并使模拟顺利进行,需要注意以下2点: (1)packmol建立的液滴模型需要在MS中加个box,即建立成“晶体”,才可以与基底合并,且“晶体”的a和b需要和基底的保持一致,即a=b=180.748。 (2)液滴的密度应用在模拟温度下的密度保持一致,300K下水的密度约为1 g/cm3。 假设液滴的半径的2.5 nm,根据密度可以算出该液滴中水分子有个。液滴在a和b方向上在基底中央,c方向上在基底表面上方5 Å,则可知液滴球心位置为:90.374 90.374 35.84488。 Packmol的输入文件如下: # #表示注释 tolerance 2.0 #tolerance表示距离约束容差,可以理解为原子间最小距离不低于该值,对于水分子和氯离子钠离子来说2Å是个不错的值 filetype pdb #filetype表示原子类型,这里选择pdb格式 output Droplet_R20.pdb #output表示输出文件,droplet.pdb为输出文件名 # 1000个水分子置于半径为20球内部 # 球心位于原点 structure H2O.pdb #以H2O.pdb为模板建立structure number 1000 #水分子数量为1000 inside sphere 90.374 90.374 35.84488 20.0 #inside sphere表示在球内部,90.374 90.374 35.84488 35.84488 为球心坐标,20.0为半径 end structure #停止使用该模板建模 运行文件并把得到的文件用MS打开,如下图所示:
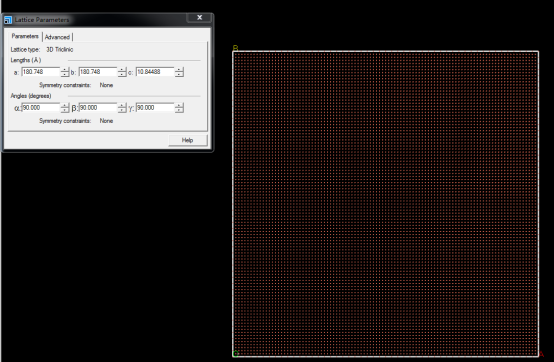
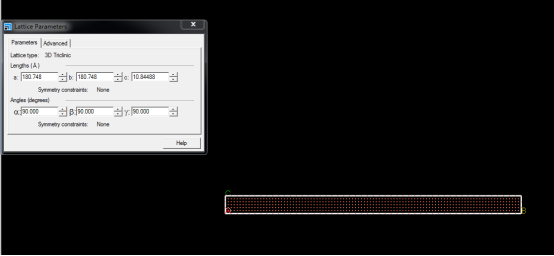
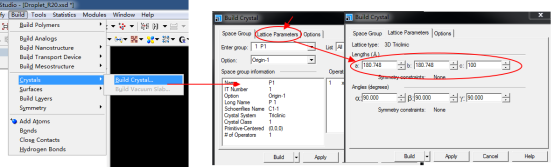
建立“晶体”,路径为:Build>crystal>build crystal并设a=b=180.748,c=100即可。
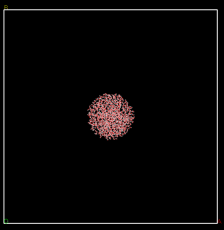
得到模型如下:
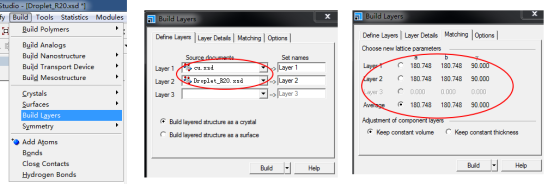
合并基底和液滴 合并液滴和基底,路径为Build>Build layers,层1为基底,层2为液滴,注意查看两个模型的a和b是否相等,不等的话最好进行调整,此处模型没有问题。
合并后模型如下,该模型经过简单的设置就可以导出进行模拟了!
感谢鲍路瑶老师的分享,内容来自于鲍老师分享出来的资料 如有需要添加微信:lmp_zhushou 进入微信群,帮助他人,共建社区 获取完整版lammps讲义可以加微信lmp_zhushou或加入QQ群994359511 |