|
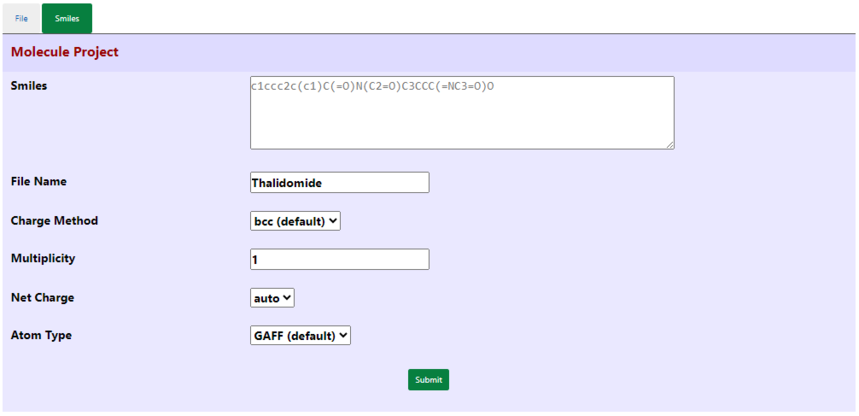
acpype在线使用https://bio2byte.be/acpype/ ACPYPE 门户旨在 为不常见的有机化合物生成拓扑参数文件。它基于 ANTECHAMBER,到目前为止,ACPYPE 是 能够为 CNS/XPLOR、GROMACS、CHARMM 和 AMBER 工作。 ACPYPE Server 的主要范围是帮助为 涉及未知参数分子的自动分子动力学模拟, 例如,在蛋白质和抑制剂的复合物中,配体通常为 一种不寻常的化合物。 ACPYPE 代表 AnteChamber PYthon Parser interfacE 的 e 和 “ace + pipe” 的发音。 引用链接 SOUSA DA SILVA, A. W. & VRANKEN, W. F. ACPYPE - AnteChamber PYthon Parser interfacE. BMC Research Notes 2012, 5:367 KAGAMI, L. P., SOUSA DA SILVA, A. W., DÍAZ, A., & VRANKEN, W. F., The ACPYPE web server for small-molecule MD topology generation, Bioinformatics, Volume 39, Issue 6, June 2023, btad350 如果您使用非均匀的 1-4 比例因子转换(例如,如果使用 GLYCAM06),请引用: BERNARDI, A., FALLER, R., REITH, D., and KIRSCHNER, K. N. ACPYPE update for nonuniform 1–4 scale factors: Conversion of the GLYCAM06 force field from AMBER to GROMACS. SoftwareX 10 (2019), 100241. 使用范围 它将采用 SMILES 输入或格式为 PDB、MDL 或 MOL2 的无开放价有机小分子的文件,并且 根据 GAFF(Amber力场)分配电荷和力场参数化。 有一些选项可以应用于提交的项目。 对于原子电荷方法,有三种方法: bcc:用于半经验性 AM1-BCC,参数化以重现 HF/6- 31G* RESP 电荷;速度慢,但具有良好的成本/收益(默认) Gasteiger 方法,非常快但不太准确 user:对于已计算电荷的 MOL2 文件,通过 R.E.D.-III 等方式 对于 net charge(净电荷),请为项目设置一个整数值,或让 ACPYPE guess 分子的净电荷。 对于 atom type(原子类型),设置 GAFF(默认)、GAFF2 或 AMBER。如果设置为 AMBER,则 ACPYPE/antechamber 将尝试根据 AMBER14SB ForceField 设置参数和 Atom 类型。 案例 失败时,将使用 GAFF 参数(但使用 AMBER 原子类型)。 不能使用:它不适用于具有开放价的有机分子;包含其他原子 比 C、N、O、S、P、H、F、Cl、Br 和 I;或共价 与另一个分子键合。 如果想要修饰氨基酸残基的参数 ,获得它的一种方法是中和 N 端和 C 端,然后拟合 手动将附加参数添加到改性残基中。 操作使用
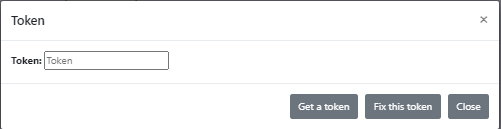
点击Token, get a token 先得到Token
此在线服务支持的文件格式为SMILES、PDB、MDL或MOL2,请求限制为200个原子。 如果可能的话,我们强烈建议您上传PDB、MDL或MOL2文件,而不是使用SMILES输入(这种表示法是分子结构的近似表示,有时会导致生成的模型不准确)。
可以批量提交
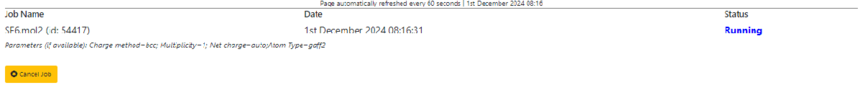
成功后
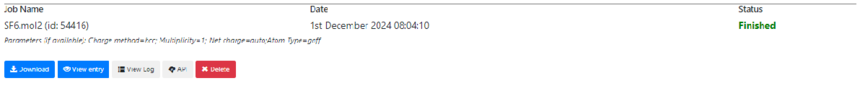
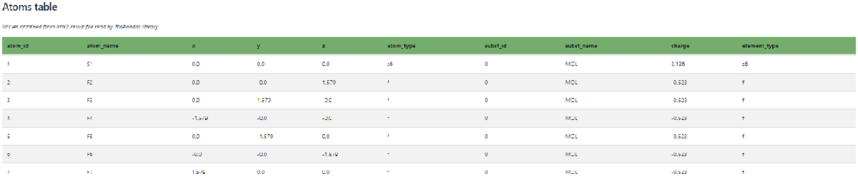
下载文件 查看项目 可以查看下载文件
Entry view
|