|
利用 SPICA力场对可离子化的脂质纳米颗粒的计算模拟研究时间:2025-07-07
*欢迎大家去观看原文,并引用 标题:In Silico Study of Ionizable Lipid Nanoparticles Using the SPICAForce Field 期刊:J. Chem. Theory Comput. 网址:https://doi.org/10.1021/acs.jctc.5c00498
一、文章摘要脂质纳米颗粒(LNPs)由可离子化的氨基脂质、磷脂酰胆碱(PC)脂质和胆固醇组成,已被证明在多种应用中(包括癌症免疫疗法、细胞重编程、基因编辑和病毒疫苗,例如针对 COVID-19疫苗)作为治疗性寡核苷酸的传递载体具有良好的前景。然而,关于可离子化的氨基脂质及其组装体(如 LNPs)的分子表征(包括在计算机模拟和体外实验中)仍处于早期阶段。特别是,由于需要精确的粗粒度(CG)模型,关于 LNPs 的计算机模拟研究以了解其纳米结构一直较为有限。在本研究中,我们扩展了 SPICA力场,以开发一个更可靠和更准确的显式粗粒度模型,用于通过计算机模拟实验研究模型 LNPs 的结构和性质。使用这个粗粒度模型,我们对具有不同辅助脂质和 pH条件的 LNP系统进行了分子动力学模拟。我们的结果表明,在 pH4时,LNPs 中的双链 DNA(dsDNA)夹在紧密接触的单层之间形成了双层结构;而在 pH7时,dsDNA 分子则嵌入到 LNPs内部的无定形区域中。这些通过计算机模拟优化的微结构与从小角度 X射线散射和低温透射电子显微镜(低温 TEM)获得的实验观察结果高度吻合。此外,对含有不同辅助脂质的脂质纳米颗粒(LNPs)的详细分析解释了为何用不饱和 PC脂质取代饱和 PC脂质会提高 DNA转染活性。总体而言,这项研究为 LNPs 的计算机模拟研究提供了可靠的 CG模型,并提供了深入的分子层面见解,以改进其设计以提高稳定性和有效性。
二、计算图文
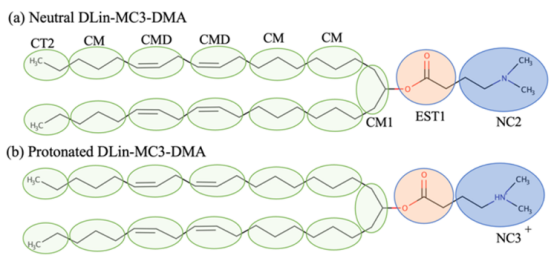
图 1.(a)未带电的 MC3 的 CG 分子模型以及(b)带正电的 MC3 的 CG 分子模型。透明的椭圆形框表示由单个 CG段所代表的原子群。
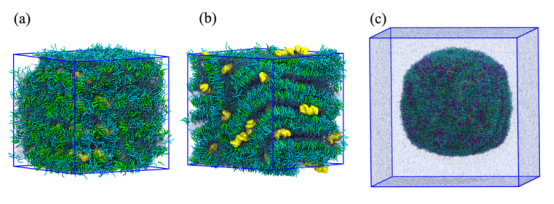
图 2.(a)阳离子型 MC3、胆固醇(CHOL)和双链 DNA 的初始配置,用水进行少量润湿处理。 (b)经过约400 纳秒的 CG模拟后系统的快照。 (c)加入磷脂(PC)并施加球形壁电位后的悬浮水中的球形脂质纳米粒。颜色方案:阳离子型 MC3为青色,胆固醇为绿色,DOPC/DSPC为紫色,双链 DNA为黄色。水用半透明的冰蓝色表示。
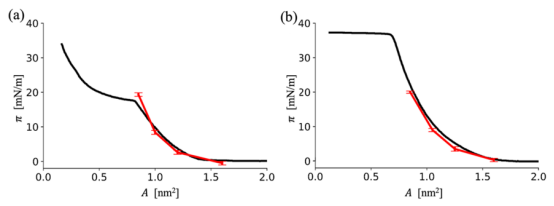
图3. (a)中性 MC3 和 (b)阳离子 MC3 在缓冲水相下表面压力(π)与分子面积(A)的关系曲线。黑色实线代表实验所得的 π - A曲线,而红色线则表示通过优化参数进行 CG MD模拟得到的 π - A曲线。
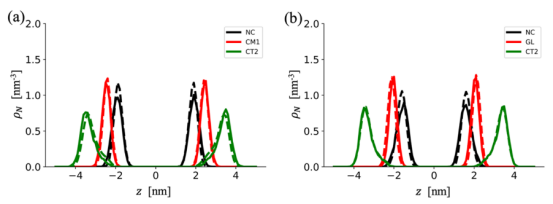
图 4 。选取的 CG片段在阳离子 MC3/DOPC二元单层表面的密度分布情况,分别对应于(a)阳离子 MC3脂质和(b)DOPC 的情况。实线表示 CG模拟的结果,而虚线则表示 AA模拟的结果。
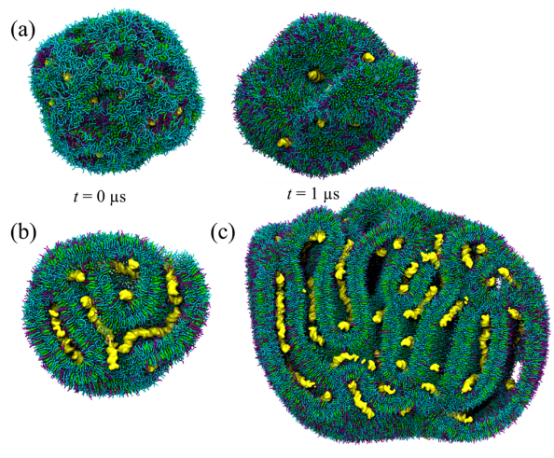
图 5.(a)基于阳离子 MC3 的 LNPs与 DSPC 在水溶液中进行的 CG MD模拟的时间演化过程。LNPs 的横截面视图展示了基于阳离子 MC3 的 LNPs 中各成分的排列情况,在(b)小长度尺度和(c)大长度尺度下均有展示。颜色方案:阳离子 MC3脂质为青色,胆固醇为绿色,DSPC为紫色,dsDNA为黄色。为清晰起见,水和离子未绘制在图中。
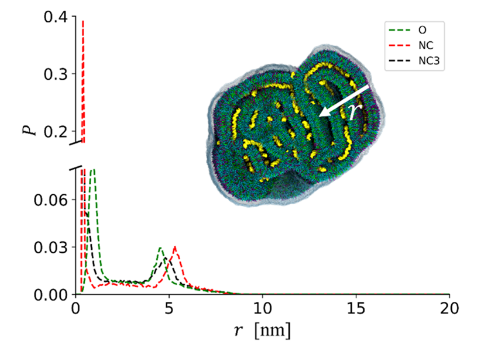
图6.以阳离子型 MC3/DSPClipid为基础的脂质纳米粒在纳米粒表面法线方向上脂质的分布情况,从纳米粒表面(r =0纳米)到纳米粒内部。颜色方案:阳离子型 MC3为黑色,DSPC为红色,胆固醇为绿色。插图:带有表面平均疏水面积(SASA)的纳米粒横截面视图。颜色方案与图2相同。
图7. 在水溶液中,基于中性 MC3 的脂质纳米颗粒在(a)小尺度和(b)大尺度下的横截面视图。颜色方案:中性 MC3脂质为青色,胆固醇为绿色,DNA为黄色,DSPC为紫色。为清晰起见,水和离子已省略。
图 8 。比较了(a)MC3脂质和(b)胆固醇在阳离子型和中性型 MC3脂质包被的脂质纳米粒(pH4 对比 pH7)表面法线方向上的概率分布情况,该分布是从脂质纳米粒表面(r =0纳米)沿其各自的中心方向进行测量的。
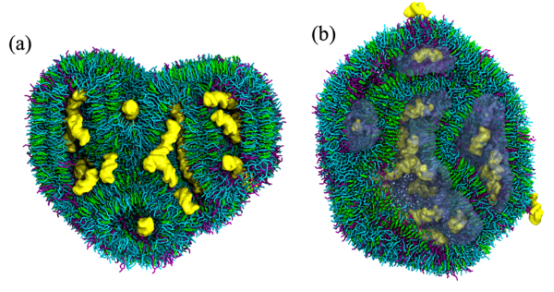
图 9.基于 MC3 的 LNPs 在 DOPC存在下的横截面视图,在水溶液中分别于(a)pH4 和(b)7时呈现。颜色方案:蓝色表示带电或中性的 MC3脂质,绿色表示胆固醇,黄色表示 DNA,紫色表示 DSPC。为清晰起见,水和离子未画出。
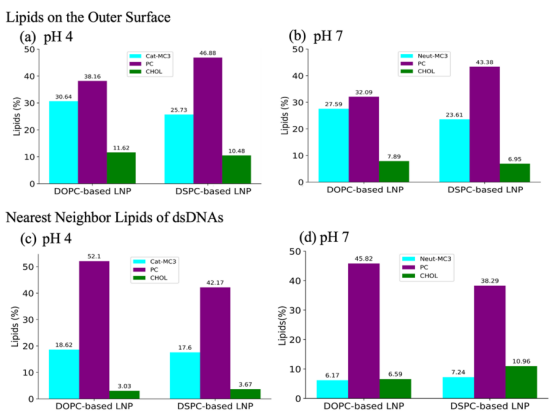
图10.含有 DOPC 和 DSPC 的脂质纳米粒表面脂质的百分比(a)在 pH约为4 的条件下,(b)在 pH约为7 的条件下;以及脂质纳米粒内部围绕 dsDNA 分子的最近邻脂质的百分比(c)在 pH约为4 的条件下,(d)在 pH约为7 的条件下。每个脂质类型的百分比均根据系统中脂质类型的总数重新计算得出。
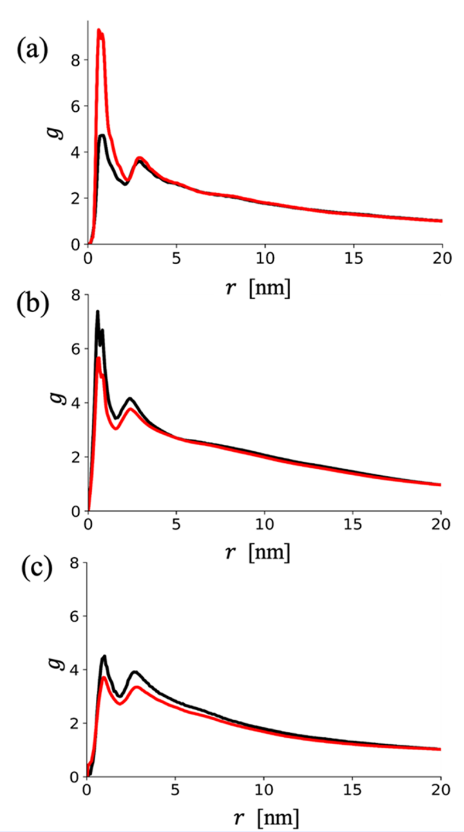
图11. RDF 图显示了(a)PClipids 在 CHOL周围的质心分布情况,(b)中性 MC3脂质在 CHOL周围的分布情况,以及(c)中性 MC3脂质在 PC周围的分布情况。颜色方案表示含有 DOPC脂质的 LNP(黑色)和含有 DSPC脂质的 LNP(红色)。
三、计算分析3.1. 模拟计算分析的主要内容文章通过CG MD模拟,聚焦于LNPs的分子级结构、动力学行为和功能机制。具体分析内容如下:
3.1.1 LNPs纳米结构的pH依赖性演化模拟揭示了LNPs在酸性(pH ~4)和生理(pH ~7)条件下的结构差异,关键发现包括: pH ~4(模拟内体条件):LNPs形成双层结构(bilayer),双链DNA(dsDNA)被夹在带正电的脂质双层之间(图5b)。这种结构源于可电离脂质(如MC3)的质子化,促进静电相互作用,导致DNA与脂质的紧密结合。模拟显示,双层厚度约为4.5 nm,与实验cryo-TEM数据一致。 pH ~7(生理条件):LNPs转变为非晶态水域结构(amorphous water domains),DNA嵌入颗粒内部的水域中(图7)。由于MC3脂质中性化,静电吸引力减弱,导致DNA分布松散,部分区域出现泄漏(尤其在小尺寸LNPs中)。这解释了LNPs在储存中的不稳定性。
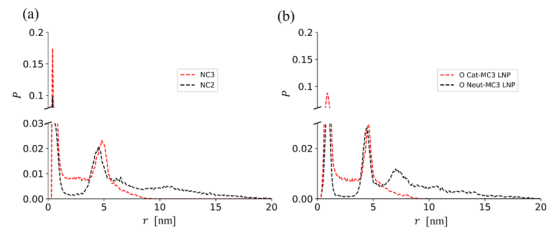
3.1.2 辅助脂质(Helper Lipids)的影响机制文章分析了饱和脂质(DSPC)与不饱和脂质(DOPC)对LNPs性能的影响: 结构差异:DOPC基LNPs的表面覆盖较差(DOPC仅占表面脂质的4.7% vs. DSPC的4.0%),导致颗粒更松散(半径回转Rg较大),而DSPC基LNPs的胆固醇(CHOL)分布更均匀(图10)。这与实验观察到的DOPC基LNPs具有更高转染活性但稳定性较低的现象一致。 分子相互作用:通过径向分布函数(RDF)分析,DSPC与CHOL的相互作用更强(RDF峰值更高),而DOPC则较弱,导致DOPC基LNPs内胆固醇溶解性差,引发结构不稳定(图11a)。
3.1.3 DNA-脂质复合物的自组装行为模拟追踪了DNA与脂质的动态结合过程: 自组装机制:在有限水条件下,阳离子MC3脂质通过静电作用驱动DNA形成层状结构(图S6)。CG模型成功复现了全原子(AA)MD观察到的自组装模式,DNA被夹在脂质双分子层之间,半径回转(Rg)误差小于0.4 Å(表S5)。 DNA分布:概率分布分析显示,在pH ~4时,DNA靠近LNP表面(r = 0–8 nm),而在pH ~7时,分布更广泛(r = 3–18 nm),证实了DNA在内部水域的嵌入(图S9)。
3.1.4 脂质分布与表面组成通过概率分布和组分分析,量化了LNPs内脂质的空间分布: 表面vs.内部脂质:在pH ~4时,小LNPs表面脂质占比22%(大LNPs为16%),而内部以MC3和CHOL为主(图6)。水体积分数为21–24%,与实验数据吻合。 核心结构:中性MC3脂质在pH ~7时优先定位到LNP核心(图8a),形成类固态结构,支持了实验中“中性脂质促进核心形成”的假说。
四、计算方法本文通过分子动力学模拟研究可电离脂质纳米颗粒(LNPs),其计算体系包含全原子(AA)模拟和粗粒度(CG)模拟两个层次,核心是扩展SPICA力场(SPICA FF)并优化参数。以下从科研角度详细拆解计算方法和参数体系,结构分为四部分:
4.1. 力场模型与能量函数4.1.1 SPICA力场扩展文章扩展了SPICA力场以支持LNPs组分,新增三种CG粒子类型: NC2(中性MC3头基:-CH₂-CH₂-N(CH₃)-CH₃) NC3(质子化MC3头基:-CH₂-CH₂-NH⁺(CH₃)-CH₃) CM1(连接基团:-CH₂-CH-CH₂-) 表1:CG粒子类型定义(原文Table 1)
4.1.2 能量函数形式总势能 Utotal 包含键合项 (Uintra) 和非键项 (Unonbond): 键合项(键长、键角、二面角):Uintra=bond∑kb(r−r0)2+angle∑kθ(θ−θ0)2+dihedrals∑kφ[1+cos(nφ−δ)] 其中 kb,kθ,kφ 为力常数,r0,θ0 为平衡值(通过AA MD拟合)。 特殊校正:为避免过度弯曲,引入1-3相互作用的LJ校正项(公式2)。
非键项:范德华作用:分两类 水相关对:LJ12-4势 其他对:LJ9-6势 静电作用:库仑势(相对介电常数 εr=80)
4.2. 参数优化策略参数通过多尺度验证优化,目标复现实验和AA MD数据:
4.2.1 键合参数优化方法:映射AA MD轨迹(CHARMM36力场),拟合键长/键角分布 示例:图S3对比AA与CG的键长(NC2-EST1、EST1-CM1)和键角(EST1-CM1-CM)分布,手动调整 kb,kθ 以匹配峰值和离散度。
4.2.2 非键参数优化表面张力(γ)匹配AA参考:模拟二元单层(如MC3/DOPC) 系统:100个MC3 + 100个DOPC,面积/脂质=0.95 nm²,水分子~1800个(表S1)。 收敛验证:图S4显示γ在100 ns内收敛。 CG优化:调整 εij,σij 使CG γ ≈ AA γ(误差<3 mN/m,表2)。
密度剖面匹配:对比AA与CG的脂质头基沿法线方向分布(如MC3的NC2与DOPC的PH),优化LJ参数(图4)。
DNA-脂质复合物Rg匹配:AA参考:模拟50个MC3 + 1个dsDNA(12 bp) 结果:中性MC3/DNA的 Rg=24.2±0.2 Å(表S5)。 CG优化:调整NC3-DNA静电参数,使CG Rg误差≤0.4 Å(表S5)。
4.2.3 实验校准π-A等温线:通过Langmuir槽实验测量MC3单层表面压力,优化CG参数复现曲线(图3)。
4.3. 模拟系统构建4.3.1 初始配置生成LNP构建流程(图2):核心自组装:随机放置阳离子MC3、胆固醇、dsDNA于小盒子(水体积分数24%),运行CG MD至自组装(400 ns)。 添加外壳:将组装体置于更大盒子,用PACKMOL添加PC脂质(DOPC/DSPC)形成球形外壳,加水至实验浓度。
关键参数:小LNP:6,400 MC3脂质 + 4,992胆固醇 + 64 dsDNA + ~415,000水粒子(表S2)。 大LNP:51,200 MC3脂质 + 39,936胆固醇 + 512 dsDNA + ~5.15×10⁶水粒子(表S2)。
4.3.2 模拟设置软件与硬件:AA MD:GROMACS 5.0.4,PME静电,截断半径1.2 nm。 CG MD:LAMMPS,P3M静电,时间步长5 fs,LJ截断15 Å。
系综控制:单层系统:NVT系综(固定盒子尺寸)。 LNP系统:NPT系综(Andersen恒压器1 atm,Nosé-Hoover恒温298 K)。
模拟时长:
4.4. 关键分析指标4.4.1 结构表征指标概率分布:沿LNP表面法线(r=0至中心)分析脂质/DNA分布(图6),定义: P(r)=总粒子数粒子数 at r(r: 距SASA表面的向内投影) 径向分布函数(RDF):量化脂质间相互作用(图11),如DSPC与CHOL的RDF峰值反映结合强度。
4.4.2 动态行为指标半径回转(Rg):追踪LNP尺寸变化(图S12) 水合数演化:监测结合水分子数验证系统平衡(图S7-S8)
|