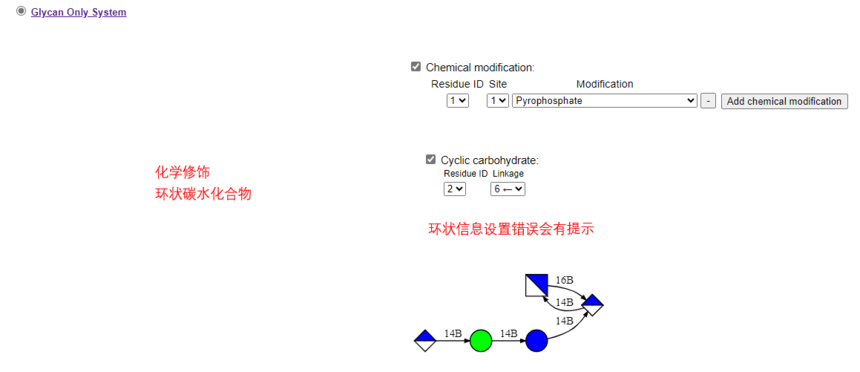
|
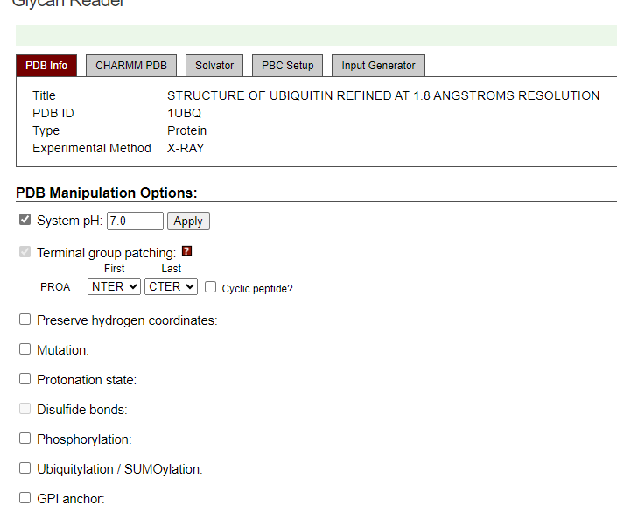
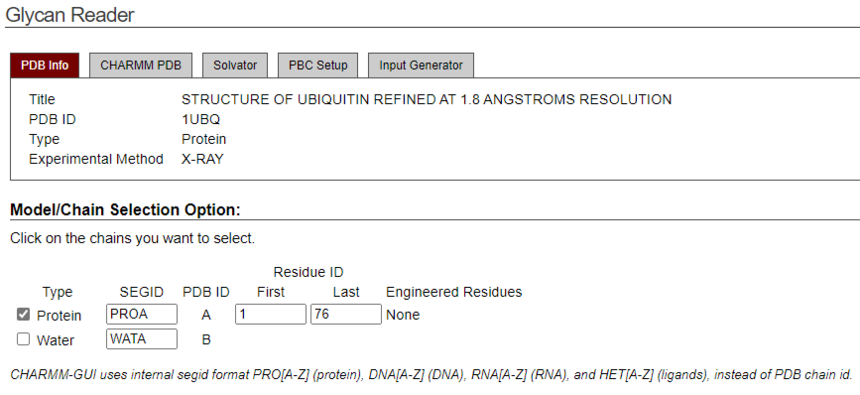
Glycan Reader & ModelerGlycan Reader&Modeler通过以下方式极大地简化了对带有聚糖的PDB结构文件的读取 自动检测类糖结构, 根据几何形状和立体化学确定和分配正确的糖类型, 糖苷键的识别和聚糖链的构建,以及 生成CHARMM输入 此外,Glycan Reader&Modeler支持通过以下方式对用户指定的N-/O-聚糖或纯聚糖结构进行建模 从GFDB和 通过糖苷角的刚体旋转寻找正确的方向 请注意 为了正确识别糖残渣名称及其链接信息,碳水化合物原子应在HETATM中列出,并带有链ID,它们的连接信息必须在PDB文件中提供。如果缺少连接信息,可以使用PyMOL生成它。PyMOL根据原子-原子距离构建CONECT表,因此请确保正确绘制所有键(或查看是否添加了不必要的键)。 Glycan Reader&Modeler嵌入在PDB Reader中,因此可在大多数其他模块中使用,例如Quick MD Simulator、薄膜生成器等。 聚糖阅读器和建模器支持PDB中糖残留物的大多数化学修饰。 •Glycan Reader&Modeler支持PDB Reader中的所有选项。 如果您不熟悉第一个PDB阅读步骤,请先观看这些视频演示。 更多聚糖相关工具可以在GlycanStructure.ORG找到。 参考文献 S. Jo, T. Kim, V.G. Iyer, and W. Im (2008) CHARMM-GUI: A Web-based Graphical User Interface for CHARMM. J. Comput. Chem. 29:1859-1865 S. Jo, K.C. Song, H. Desaire, A.D. MacKerell, Jr., and W. Im (2011) Glycan Reader: Automated Sugar Identification and Simulation Preparation for Carbohydrates and Glycoproteins. J. Comput. Chem. 32:3135-3141 S-J. Park, J. Lee, D.S. Patel, H. Ma, H.S. Lee, S. Jo, and W. Im (2017) Glycan Reader is Improved to Recognize Most Sugar Types and Chemical Modifications in the Protein Data Bank. Bioinformatics 33:3051-3057 S-J. Park, J. Lee, Y. Qi, N.R. Kern, H.S. Lee, S. Jo, IS Joung, K. Joo, J. Lee, and W. Im (2019) CHARMM-GUI Glycan Modeler for Modeling and Simulation of Carbohydrates and Glycoconjugates. Glycobiology 29:320-331 第1步 输入蛋白id
第2步 处理蛋白的截断序号
第3步 处理蛋白的改性
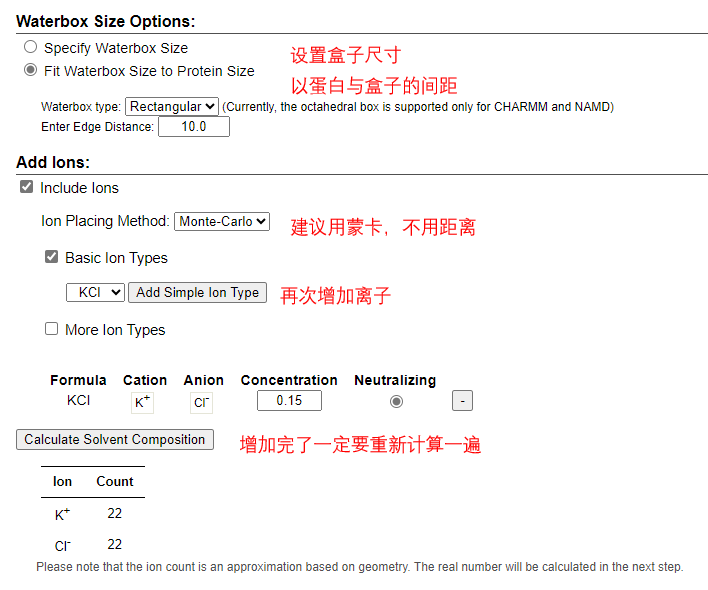
第4步 设置盒子大小 添加离子
更多离子包含 碱金属 碱土金属 过渡金属 后过渡金属 稀土元素 放射性元素 多原阳离子 多原子阴离子 卤素 第5步 设置FFT(快速傅里叶变换)
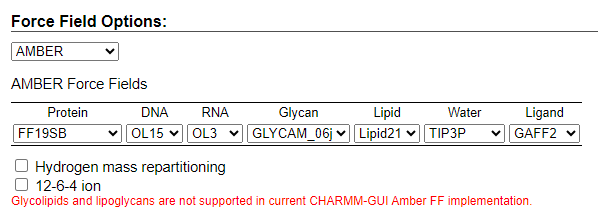
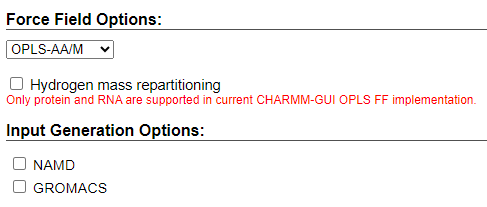
第6步 输出需要软件的计算参数 可用力场
CHARMM36m:是 CHARMM36 的改进版本,主要针对折叠和内在无序蛋白质体系进行了优化,在生成内在无序肽和蛋白质的多肽主链构象系综方面有更高的准确性
第6步 第一个是加了一些nbfix pair,来做cation-pi interaction。主要就是优化了WYF氨基酸和阳离子之间的作用 氢质量分配(HMR)的意义在于提高分子动力学(MD)模拟的效率和覆盖相空间的能力。 传统的全原子MD模拟由于需要较小的积分时间步长(通常为1或2飞秒)来保持总能量的守恒,导致相空间的覆盖速度较慢, 限制了其在缓慢演化系统中的应用。HMR技术通过将重原子的质量分配给与其相连的氢原子,使得可以使用更大的时间步长(如4飞秒),从而提高了模拟的效率。 就是用超过一个点,来描述钙离子,更准确描述钙离子和蛋白质的相互作用,类似于TIP5P的这种方式 选择AMBER力场
当前的CHARMM-GUI Amber FF实现不支持糖脂和脂多糖。 选择OPLS力场
当前的 CHARMM-GUI OPLS FF 实现仅支持蛋白质和 RNA。 仅Glycan
|


 】
】