|
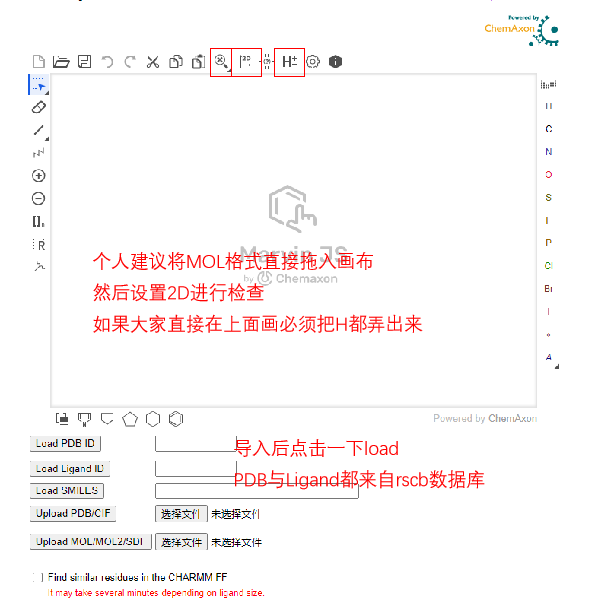
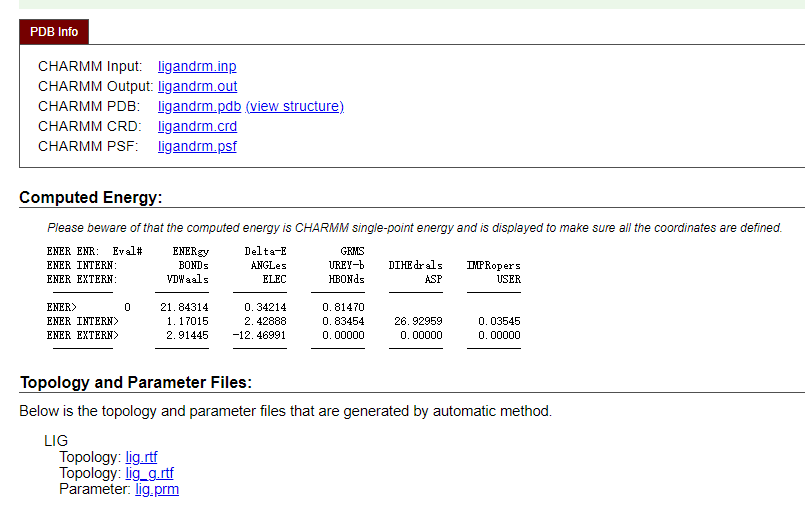
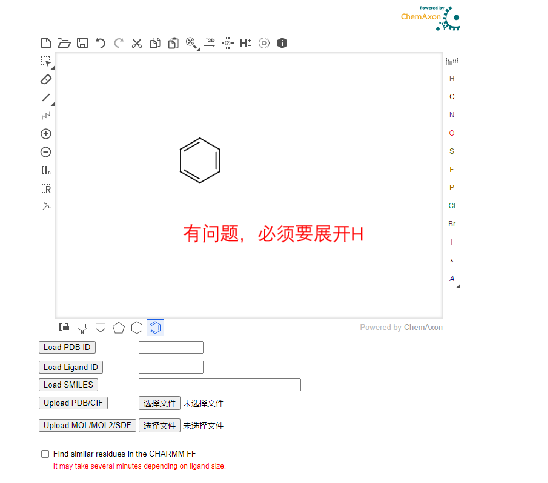
Ligand Reader & Modeler配体阅读器和建模器旨在提供: •如果定义了配体,则CHARMM拓扑文件中的残差名称, •如果没有,使用CgenFF的配体的CHARMM兼容拓扑和参数文件 •配体的PSF/CRD/PDB文件 因此,这些输出可用于其他CHARMM-GUI模块,以对含有配体的生物分子系统进行建模。 请注意 视频演示中有一个配体阅读器和建模器演示来说明如何使用这个模块。 此页面由Marvin JS提供支持。请参阅其用户指南以获取帮助。 仅识别PDB文件中HETATM记录中的分子。 由于碳水化合物通过聚糖阅读器在PDB阅读器中自动识别,因此不处理碳水化合物。 当PDB文件中有多个相同配体的分子时,只有一个结构用于输入生成。 配体结构及其拓扑和参数文件基于Marvin JS上显示的结构。因此,请确保Marvin JS结构正确,包括手性和键序。 •在“Load PDB ID”和“Load Ligand ID”的情况下,根据所选配体的RCSB SDF文件显式生成氢原子。在“Upload PDB File”的情况下,配体阅读器和建模器检查所选配体是否在RCSB化学成分字典中可用。如果所选配体结构与RCSB SDF结构的结构或子结构相同,则从SDF结构中取出氢原子;否则,根据原子类型生成以填充“隐式价”。用户可以在必要时修改质子化状态。 所有氢原子和缺失的原子都应该明确放置,以获得准确的结果。 如果使用Marvin JS修改上传的结构,则未修改的重原子的坐标将被保留。 请使用提供的选项或右键详细绘制配体,包括手性、键类型和原子电荷。 对于类脂分子(具有长酰基链),使用最大公共子结构的类似结构搜索将是不可能的。 组合结构生成是可能的。请参阅R-Group。 如果PDB结构具有多个链,未修改的链将与修改的配体组合到PDBID_modified. pdb文件中。该文件可用于其他CHARMM-GUI模块。 参考文献 S. Jo, T. Kim, V.G. Iyer, and W. Im (2008) CHARMM-GUI: A Web-based Graphical User Interface for CHARMM. J. Comput. Chem. 29:1859-1865 S. Kim, J. Lee, S. Jo, C.L. Brooks III, H.S. Lee, and W. Im (2017) CHARMM-GUI Ligand Reader and Modeler for CHARMM Force Field Generation of Small Molecules. J. Comput. Chem. 38:1879-1886 第1步 画分子
第2步
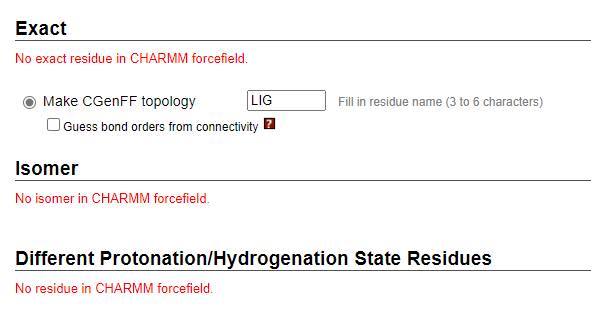
猜测键序(CGENFF) 以下是ParamChem项目网站(http://www.paramchem.org)对该选项的描述。 已经引起了我们的注意,一些流行的开源分子编辑器(最值得注意的VMD)产生了具有无效键序的mol2文件。此外,扭曲的pdb经常被提交,导致OpenBabel错误地猜测键序。为了解决这些问题,新版本的界面具有一个复选框,指示程序丢弃输入键序并从连接中猜测键序。我们想强调的是,此功能应与EXTREME CAUTION一起使用!通常,程序会检查输入键序是否与连接一致,如果不是这种情况,则返回错误。这样做已经发现了许多问题,否则会导致不正确的参数。但是,通过选择“猜测键序…”来禁用此检查这意味着,例如,如果氢原子丢失或放置错误,程序可能会在没有警告的情况下输出非常不准确的参数。此外,猜测键顺序可能并不总是适用于第三行和更重的元素。 第3步
第1步
第2步
|