|
6.3选择方法时间:2025-06-14 VMD有一个相当强大的原子选择语言可用。它基于这样的假设:每个原子都有一组与其相关的值,这些值可以通过关键字访问。 这些值可以是布尔值(这是一个蛋白质原子吗?)、数字(如原子索引或原子质量)或字符串(原子名称)。这些值甚至可以通过Tcl数组来引用。 首先,这里有一些VMD中有效选择命令的示例。接下来将更深入地描述选择是如何工作的。 name CA resid 35 name CA and resname ALA backbone not protein protein (backbone or name H) name ’A 1’ name ’A *’ name "C.*" mass < 5 numbonds = 2 abs(charge) > 1 x < 6 and x > 3 sqr(x-5)+sqr(y+4)+sqr(z) > sqr(5) within 5 of name FE exwithin 3 of protein protein within 5 of nucleic same resname as (protein within 5 of nucleic) protein sequence "C..C" name eq $atomname
有两种选择模式。第一个是一个关键字,后面跟着一个值列表或一个值范围。例如, name CA 选择所有名称为CA的原子(可以是Cα或钙); resname ALA PHE ASP 选择丙氨酸、苯丙氨酸或天冬酰胺中的所有原子; index 5 选择第六个原子(在内部VMD编号方案中)。 VMD还可以进行范围选择,类似于X-PLOR的‘:’表示法: mass 5 to 11.5 选择质量在5到11.5之间的原子, resname ALA to CYS TYR 选择丙氨酸、精氨酸、天冬氨酸、天冬氨酸、胱氨酸和酪氨酸中的原子。
关键字选择通过检查关键字后面列表中的每个词来工作。术语可以是单个单词(例如,名称CA),也可以是一个范围(例如,从35到90)。 用于确定范围检查的方法从关键字数据类型确定;数值比较不同于字符串比较。比较应该按预期工作,以便“8”在数字上下文中介于“1”和“11”之间,而不是在字符串上下文中。这可能会导致一些特殊的问题。一些关键字,如segname,可以接受字符串值,但也可以被一些人用作数字字段。假设有人用数字1到12标记了段名字段,假设它们是数字。如果发现段名1到11只返回两个段,这个人可能会很困惑。此外,字符串将被转换(通过atof())为数字,因此如果字符串不是数字,它将被赋予值0。 可以使用第6.3.6节中讨论的关系操作符强制在字符串或数字上下文中进行搜索,选择可以与布尔操作符and和or组合,在括号内收集,并通过not修改,如 (name CA or name CB) and mass 12 to 17 它选择所有命名为CA或CB的原子,质量在12到17 amu之间(这可以用来区分Cα和钙)。VMD具有类似于C的运算符优先级,因此将括号放在前面的表达式之外,如: name CA or name CB and mass 12 to 17 实际上会选择所有的CA原子或CB原子,它们有合适的质量。
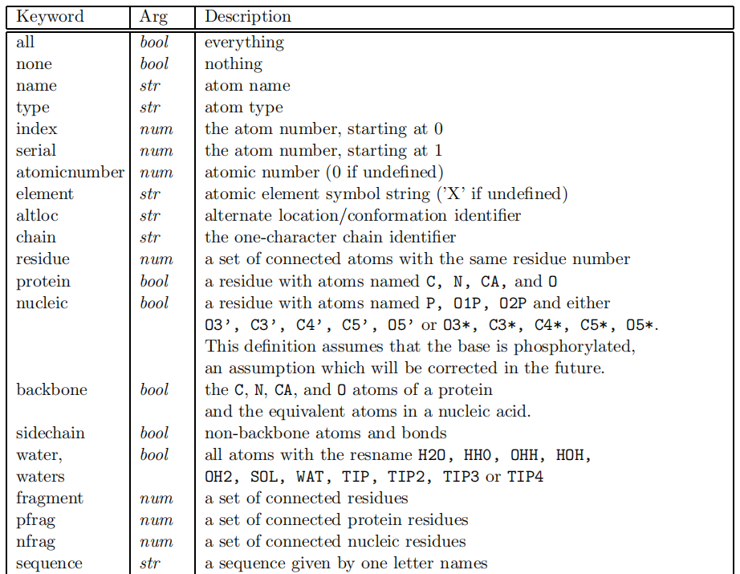
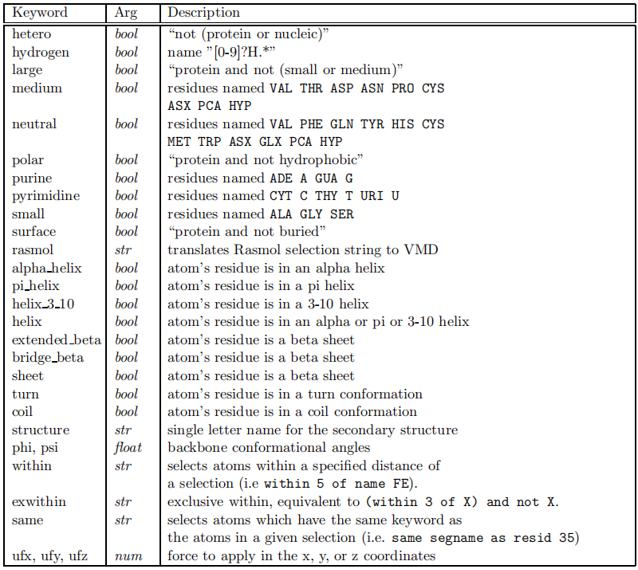
关键字和函数的定义可用于在VMD中选择原子的关键字列在本章末尾的表6.5和表6.6中。如果关键字定义后跟bool,则它要么为on,要么为off。如果后跟str,则接受字符串上下文中的值。如果后跟num,则接受数字上下文中的值。 表6.7列出了可以在带有数字关键字的原子选择表达式中使用的内置函数。 表6.8列出了内置的原子选择关键字,可以在原子选择表达式中使用这些关键字来查询同一分子中底层体积图的值。这些是只读的。
布尔的关键词有些选择不带值。例如,主链选择蛋白质的主链原子,核酸和蛋白质选择蛋白质原子。为这些选择提供选项是错误的。 这些选择可以像其他选择一样使用,如下所示: protein and backbone nucleic or protein
此外,如果neither和nor或位于布尔关键字之后,则插入隐式的and,从而使以下内容有效: protein name CA (same as: protein and name CA) nucleic backbone
Short CircuitingVMD中的布尔逻辑在元素的基础上进行短路计算。例如,给定一个原子,如果X为真,那么无论Y的值如何,X或Y都会为真,因此不需要对其求值。类似地,如果X为假,那么X和Y也将为假,因此Y不需要再次求值。 了解短路选择的工作原理可以加快几种类型的选择。考虑一个含有大量水和蛋白质的系统。表达protein和segname < 10比表达segname < 10和protein要快,因为在第一次选择中,只有作为蛋白质的原子将segname转换为数字,而在第二次选择中,所有的片段名称都被转换。 内选有其自身的短路形式。该命令可以被解释为“找到A中距离B给定距离内的原子”,如果A没有给定,则搜索所有原子。在VMD中进行搜索所需的时间大致与a中的原子数乘以B中的原子数成正比,因此减少a中的原子数(即,不测试每个原子)可以使搜索更快。 使用该系统与大量的水和蛋白质,比较选择 protein within 5 of resid 1 to (within 5 of resid 1) and protein. 第一种方法非常快,因为它在所有蛋白质原子和残渣1中的所有原子之间进行距离搜索。然而,第二次选择在所有的渣滓5˚A范围内的原子中搜索,然后找出其中哪些是蛋白质原子。
单引号引用VMD允许两种引用机制,单引号和双引号。单引号用于包含空格和其他非字母数字字符。信不信由你,有一些残留的名字里面有一个空格,所以它们可以被引用为,例如, resname ’A 1’ 更重要的是,核糖原子可以被命名为C5 '或C5*(取决于PDB记录的年龄)。VMD中的词法分析器已被修改,因此可以使用不带引号的C5 ‘, O ’和N ',但它不能处理未加引号的星号(*与乘法冲突,解析器无法解决差异)。一些例子是: name ’O5*’ segname ’A *’ name O5’ 引号也可以用来绕过保留的选择词,比如x。选择命令segname x会给出一个错误,因为x是另一个关键字。相反,使用段名‘ x ’。有一种转义机制可以在单引号字符串中包含单引号,该机制在单引号之前使用反斜杠(' \ ')。这允许不寻常的名称,如C ‘被引用为’ C \ "。 segname x <---- error; conflicts with the ’x’ keyword segname ’x’ name ’O5\’’ 此外,还可以使用双引号(将在下一节讨论),如“C '”或“C \*”。
双引号和正则表达式字符串周围的双引号用于指定正则表达式搜索(与Perl 5.005兼容,使用Philip Hazel编写的Perl兼容正则表达式库)。如果您不知道如何使用它们,请尝试在手册页中查阅ed、egrep、vi或regex。如果没有,请阅读Perl文档,或者从包括O 'Reilly and Associates的Sed和Awk书籍在内的众多书籍中选择一本。下面的示例仅展示了在VMD中使用正则表达式的几种方式。
选择所有名称以C开头的原子: name "C.*" 包含数字的段名: segname ".*[0-9]+.*" 在匹配的关键字列表中可以提供多个术语。这个例子选择以A开头的残基,甘氨酸残基和以t结尾的残基。对于字符串,数字上下文中的正则表达式将被转换为整数,该整数始终为零: resname "A.*" GLY ".*T" 包含特殊字符(如+、−或*)的选择必须用\字符转义。为了选择命名为Na+的原子,可以这样选择: name "Na\+"
简而言之,常规选择允许匹配多种可能性,而不仅仅是一个字符。表6.9显示了一些可以使用的方法。 有很多方法可以做一些选择。例如,选择名称为CA或CB的原子可以通过以下方式完成: name CA CB name "CA|CB" name "C[AB]" name "C(A|B)" 对于那些已经理解正则表达式的人,有几个警告。VMD会自动在选择字符串前加上“^(”和“)$”。这使得选择O只匹配O而不匹配OG或PRO。另一方面,将^和$放入命令中不会真正影响任何东西,匹配子字符串的选择必须在前面和后面加上"。*”,比如。一些非法的选择可以被认为是正确的,但很奇怪,比如C)|(O),它被转换成^(C)|(O)$,并匹配任何以C开头或以O结尾的内容。 正则表达式类似于X-PLOR中的通配符匹配。表6.10列出了从X-PLOR样式的通配符到匹配的正则表达式的转换。
比较选择可以在VMD中使用比较来进行原子选择,例如质量< 5,选择质量小于5amu的原子,以及名称eq CA,这是选择CA原子的另一种方式。比较选择的基本思想也是基于这样一个概念,即每个原子都有一个由关键字指定的属性。当在表达式中给出关键字时,将构造对应值的数组(或向量),数组的大小与分子中的原子数相同。(如果给出单个数字或字符串而不是关键字,则数组由该给定值的副本组成。)这些操作,如加法、乘法、字符串匹配和比较,然后沿着数组逐个应用元素。这种类型的选择类似于X-PLOR中的向量语句。 以质量< 5的水为例,它有一个质量为15.9994的氧和两个质量为1.008的氢。VMD看到关键字质量,构造数组[15.9994,1.008,1.008],然后看到“5”,构造数组[5,5,5]。然后,它比较数组的每个项并返回布尔数组[False, True, True](因为15.9994不小于5,但1.008小于5)。然后使用最后的布尔数组来确定选择哪些原子;在这个例子中,是氢。 可以通过算术运算或使用一些标准数学函数(表6.7列出了这些函数)来构造更复杂的比较选择。最常用的函数可能是sqr,它将数组的每个元素平方。因此,选择空间中某点(x,y,z) =(3,4,-5)的5˚A范围内的所有原子的命令为: sqr(x-3)+sqr(y-4)+sqr(z+5) <= sqr(5)
sequenceVMD支持使用sequence selection关键字基于单字母氨基酸序列进行选择。这允许对表单进行选择 sequence APD sequence "C..C" (might be used to pick out zinc fingers) sequence AATCGGAT 不像其他字符串选择命令(接受三种类型字符串中的一种),序列的所有字符串都被作为正则表达式(尽管非字母数字字符串仍然必须加引号才能通过输入解析器)。该方法的工作原理是依次提取每个蛋白质和核酸片段(prag和nfrag),并构建一个字母的氨基酸序列。如果正则表达式匹配序列中的任何一个,则选择匹配残基中的原子。允许多个匹配,但不能重叠。与常规正则表达式一样,会进行最大可能的匹配,因此要小心使用C *C这样的表达式。
within and sameVMDare中有两种有用的选择机制:在的内和与相同的。第一个函数从一个选择中选择指定距离(in˚A)内的所有原子,包括选择本身。因此,命令: within 5 of name FE 选取FE原子5˚A以内的所有原子。该命令的一个常见用途是限制屏幕上显示的原子区域。另一个是寻找可能参与相互作用的原子。 例如: protein within 5 of nucleic 找到核酸附近的蛋白质原子。有些选择可以通过短路加速[§6.3.3]。一个相关的原子选择构造是exwithin是‘ exclusive within ’的缩写。原子选择(within 3 of protein) and not protein等价于exwithin 3 of protein same <keyword> as <selection>查找与选择中的原子具有相同“关键字”的所有原子。这可以用于选择,如same fragment as resid 35 找到所有附着在id为35的残基上的原子。可以使用任何关键字,所以选择像same resname as (protein within 5 of nucleic) 都很好,虽然很奇怪。这个命令最有用的关键字可能是residue,所以您可以说same residue as ....
Finding contact residues假设你想观察A中与B接触的原子。使用within of 选择命令。为便于演示,设A为蛋白质,B为核酸,将接触定义为A中的一个原子与B中的一个原子在2˚A以内,则选择命令为within<distance> of <selection> protein within 2 of nucleic 如果你想知道A的所有残基至少有一个原子与B接触,用same residue as (protein within 2 of nucleic)
|