|
Bilayer Membrane介绍 膜生成器帮助用户生成构建用于分子动力学模拟的蛋白质/膜复合物所需的一系列CHARMM输入。下面给出了每个步骤的简要描述。在各种其他构建方案中,用户可以在步骤3中选择“插入”或“替换”方法。(用户可以在步骤3中选择其中之一,见下文)。 ■插入方式 将蛋白质插入具有大小与蛋白质大小相当的孔的预平衡脂质双分子层中(脂质双层的文库可在档案中获得) ■更换方式 蛋白质首先由脂质样球体包装,其位置随后用于从库中随机选择脂质分子(脂质分子库可在档案中获得) 注意事项
参考文献 S. Jo, T. Kim, V.G. Iyer, and W. Im (2008) CHARMM-GUI: A Web-based Graphical User Interface for CHARMM. J. Comput. Chem. 29:1859-1865 S. Jo, T. Kim, and W. Im (2007) Automated Builder and Database of Protein/Membrane Complexes for Molecular Dynamics Simulations. PLoS ONE 2(9):e880 S. Jo, J.B. Lim, J.B. Klauda, and W. Im (2009) CHARMM-GUI Membrane Builder for Mixed Bilayers and Its Application to Yeast Membranes. Biophys. J. 97:50-58 E.L. Wu, X. Cheng, S. Jo, H. Rui, K.C. Song, E.M. Dávila-Contreras, Y. Qi, J. Lee, V. Monje-Galvan, R.M. Venable, J.B. Klauda, and W. Im (2014) CHARMM-GUI Membrane Builder Toward Realistic Biological Membrane Simulations. J. Comput. Chem. 35:1997-2004 J. Lee, X. Cheng, J.M. Swails, M.S. Yeom, P.K. Eastman, J.A. Lemkul, S. Wei, J. Buckner, J.C. Jeong, Y. Qi, S. Jo, V.S. Pande, D.A. Case, C.L. Brooks III, A.D. MacKerell Jr, J.B. Klauda, and W. Im (2016) CHARMM-GUI Input Generator for NAMD, GROMACS, AMBER, OpenMM, and CHARMM/OpenMM Simulations using the CHARMM36 Additive Force Field. J. Chem. Theory Comput. 12:405-413 J. Lee, D.S. Patel, J. Ståhle, S-J. Park, N.R. Kern, S. Kim, J. Lee, X. Cheng, M.A. Valvano, O. Holst, Y. Knirel, Y. Qi, S. Jo, J.B. Klauda, G. Widmalm, and W. Im (2019) CHARMM-GUI Membrane Builder for Complex Biological Membrane Simulations with Glycolipids and Lipoglycans. J. Chem. Theory Comput. 15:775-786 J. Lee, M. Hitzenberger, M. Rieger, N.R. Kern, M. Zacharias, and W. Im (2020) CHARMM-GUI supports the Amber force fields. J. Chem. Phys. 153:035103 S. Park, Y.K. Choi, S. Kim, J. Lee, and W. Im (2021) CHARMM-GUI Membrane Builder for Lipid Nanoparticles with Ionizable Cationic Lipids and PEGylated Lipids. J. Chem. Inf. Model. 61:5192-5202 第1步
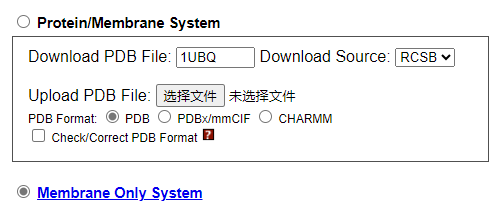
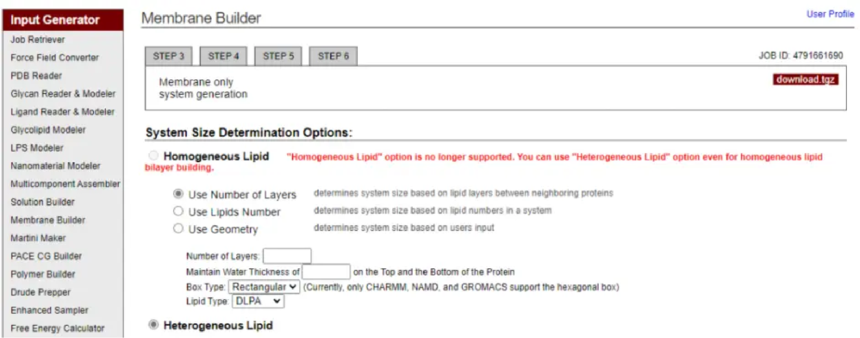
第一个跟之前一样 需要选择第二个 第2步 2)点击Next Step: Select Lipids,就会出现下图所示页面 出现一行红字:”Homogeneous Lipid”选项不在支持 可以使用”Heterogeneous Lipid”来构建Homogeneous Lipid。所以我们直接
3)点击Heterogeneous Lipid,出现下图:
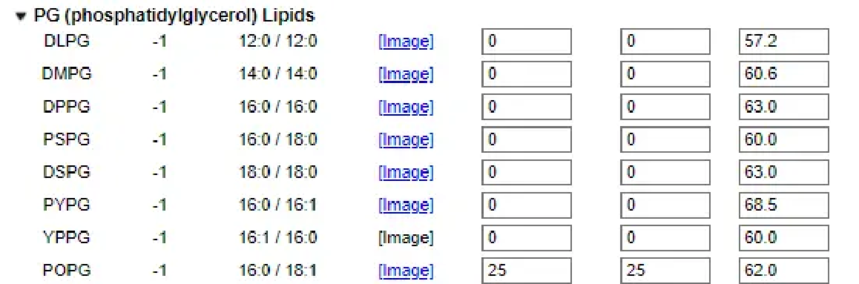
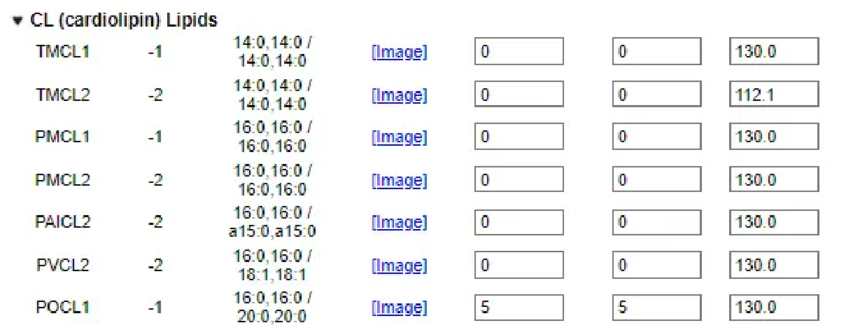
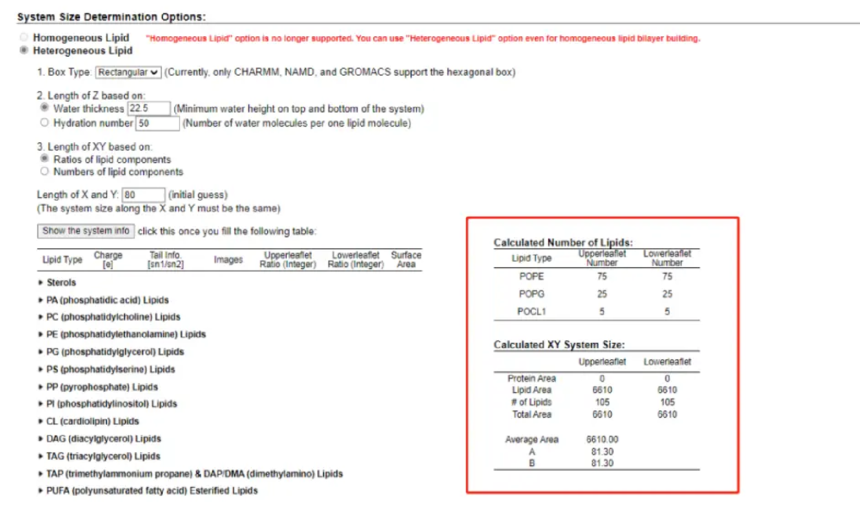
Box Type: Rectangular 长方形(默认)/Hexagonal 六边形(只有charmm、NAMD、Gromacs支持) Length of Z based on: Water thickness 水层厚度,默认值22.5,是体系顶部和底部的最小水层高度 Hydration number 水合数,默认值50,是每个脂质分子对应的水分子数 一般选择water thickness的默认值或者修改默认值 Length of XY based on: Rations of lipid components 脂质成分比率 Numbers of lipid components 脂质成分的数量 Length of X and Y : NUM (初猜)(X轴和Y轴的系统尺寸必须相同) Lipid Type 脂质类型 Charge [e] 脂质所带电荷 Tail Info[sn1/sn2] 尾信息 Images 鼠标箭头放上去就会出现脂质的二维图 Upperleaflet Ratio (Integer) 上半层脂质所占比例(具体数目) Lowerleaflet Ration(Integer) 下半层脂质所占比例(具体数目) Surface Area 脂质的表面积 其中Upperleaflet Ratio (Integer)/Lowerleaflet Ration(Integer)填写比例还是具体脂质分子数取决于 3 中,我们选取的是Rations of lipid components,还是Numbers of lipid components。 实际构建大肠杆菌细胞膜,磷脂双分子层组成:POPE:POPG:POCL1=75%:25%:5%,选择如下图:
按照百分比选择后,点击 show the system info 按钮,就会出现下图红框中的内容:
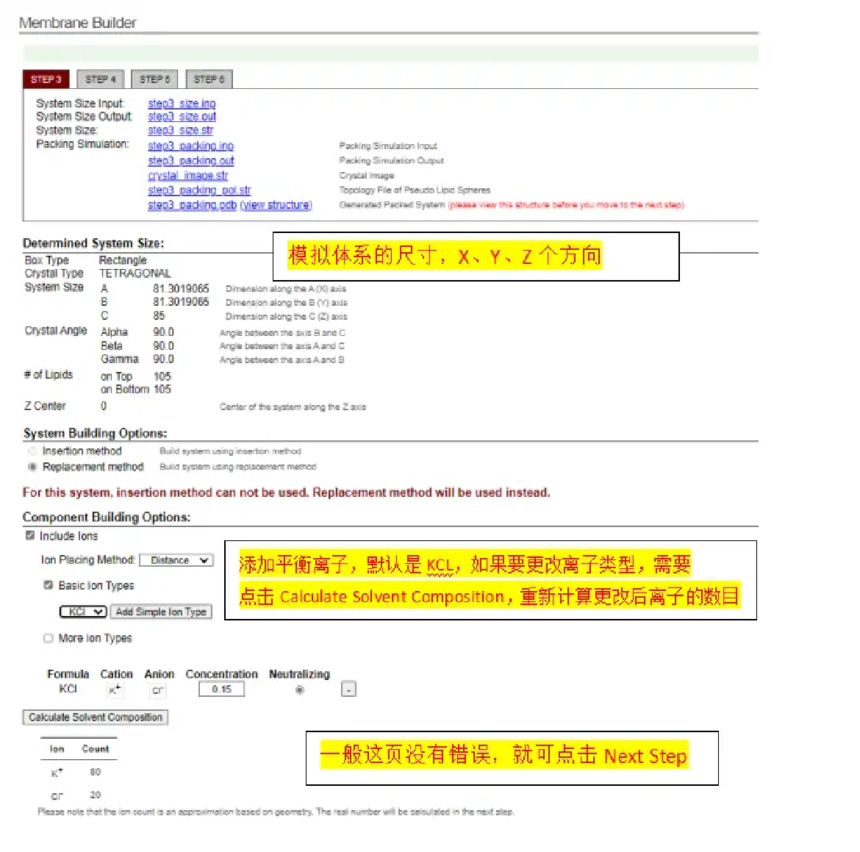
第3步 点击网页右下角Next Step: Detemine the system size,等待计算过程完成,会出现下图:
第4步 一般这页没有错误,就可点击Next Step,然后添加平衡离子,默认是KCL,如果要更改离子类型,需要点击Calculate Solvent Composition,重新a计算更改后离子的数目模拟体系的尺寸,X、Y、Z个方向,点击Next Step :Build model,等待计算完成得到下图页面:
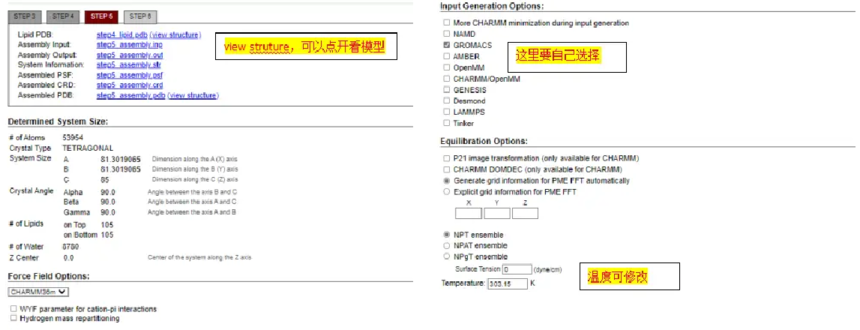
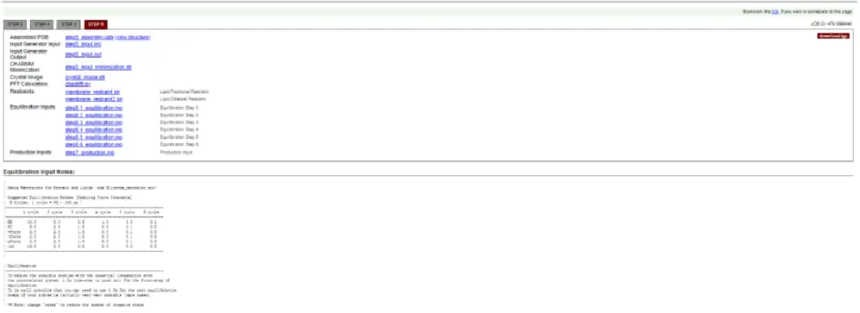
第5步
温度可修改view struture,可以点开看模型,这里要自己手动选择,选择完成后 点击Next step: Generate Equilibration and Dynamics inputs,计算完成后出现下图: 点击页面右上角的download.tgz,就能下载CHARMM-GUI建完后的非均质磷脂双分子层模型。
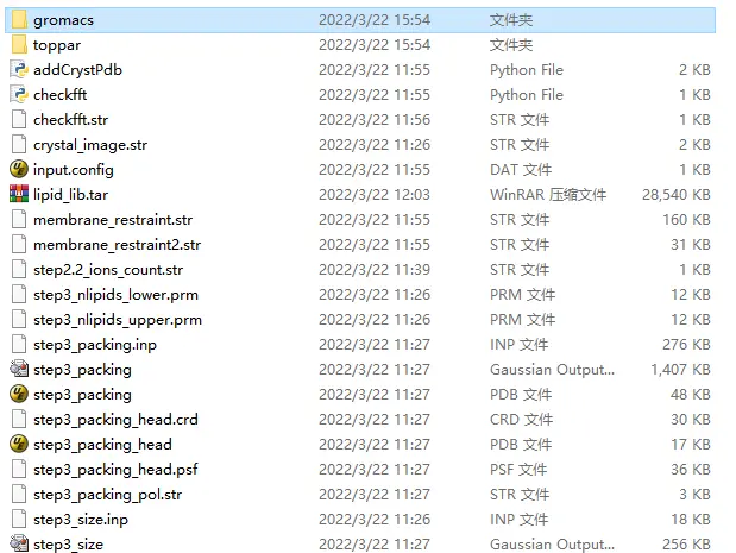
第6步 下载完成后,解压后会得到gromacs、toppar两个文件夹以及若干文件,gromacs文件夹下包含拓扑文件夹以及用于模拟的输入文件以及参数文件。
|