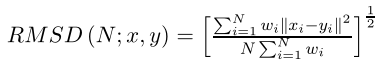|
12.4RMS配合和校准时间:2025-06-19 当一个人有两个相似的结构时,他经常想要比较它们。两种x射线结构的区别是什么?在模拟过程中,结构改变了多少?要回答这些问题,首先必须弄清楚如何比较两种结构,这通常意味着必须找到均方根偏差(RMSD)。 形式上,给定结构x中的N个原子位置和结构y中对应的N个原子的权重因子w (i),则RMSD定义为:
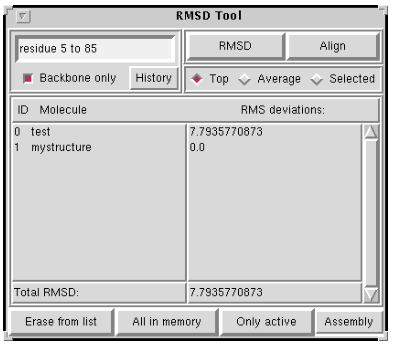
单独使用这个方程可能不会得到你想要的答案。想象两个相同的结构,相距一定距离。RMSD应该是0,但是偏移量阻止了这种情况的发生。你真正想要的是两个给定结构之间的最小RMSD;最合适的。有很多方法可以做到这一点,但对于VMD,我们实现了Kabsch (Acta crystal)方法。(1978) A34, 827-828或参见VMD源代码中的Measure.C文件)。该算法计算转换,需要将一个结构移动到另一个结构上,以最小化RMSD。 有了数学上的先决条件,我们仍然需要能够指定如何选择要比较的原子。如果你想比较两种结构中的所有原子,它们都有相同数量的原子,那么问题就很简单了,N代表一切。这种情况在MD模拟中最常见,因为两个结构之间唯一的区别就是坐标。 但是同源序列呢?在这种情况下,原子数不同是因为残基数相同,侧链的原子数不同。通常的解决方案是仅基于主链原子来确定RMSD,或者在某些仅确定了Cα原子的x射线结构中,基于Cα原子来确定RMSD。VMD允许您基于任何有效的原子选择进行拟合和对齐,只要原子选择指定要比较的每个分子中的原子数量相同。 要开始使用RMS拟合和对齐,请从扩展菜单中打开RMSD项。 您现在应该有一个名为RMSD Tool的新窗口。我们将首先描述RMSD计算器功能。
RMSD calculationRMSD计算器按钮用于计算分子间的RMS距离。菜单的左上角是指定要在计算中使用哪些原子的地方。在输入字段中,键入原子选择文本,就像在图形窗口中一样。标题为Backbone的输入字段下面的复选框只将您键入的任何原子选择限制为所选的主原子;实际上,它将“and backbone”添加到原子选择文本中。 菜单的右上角有一个标签为RMSD的按钮。其效果取决于选择了Top、Average或Selected单选按钮中的哪一个。如果选择Top, VMD计算顶部分子(通常是最后装载的分子)与其他分子之间的均方根距离。如果选择Average, VMD首先计算每个分子中所选原子的平均x、y、z坐标,然后计算每个分子到该平均结构的均方根距离。 每种分子的均方根计算结果显示在菜单下半部分的浏览器中。请注意,这个列表不会更新,直到你按下RMSD按钮,所以效果加载/删除分子不会立即反映。菜单底部的总RMSD标签显示列出的所有分子的平均RMSD。
RMS AlignmentRMS校准按钮根据选定的原子组来匹配分子。RMSD计算器按钮在不干扰分子坐标的情况下找到分子之间的RMS距离,而RMS对齐按钮实际上是将分子移动到新的位置。 这个按钮非常简单:在输入字段中输入一个原子选择,然后按Align以根据该选择中的原子对分子进行对齐。如果你用RMSD计算器按钮重新计算分子之间的RMSD,你可能会发现数值是不同的;这是因为计算是根据原子的当前位置进行的。 RMS校准按钮根据选定的原子组来匹配分子。RMSD计算器按钮在不干扰分子坐标的情况下找到分子之间的RMS距离,而RMS对齐按钮实际上是将分子移动到新的位置。 这个按钮非常简单:在输入字段中输入一个原子选择,然后按Align以根据该选择中的原子对分子进行对齐。如果你用RMSD计算器按钮重新计算分子之间的RMSD,你可能会发现数值是不同的;这是因为计算是根据原子的当前位置进行的。
RMS和脚本可以在脚本级别上执行相同的操作。Text界面还通过允许选择适合/比较的原子的原子选择机制为您提供了更大的灵活性。
RMSD Computation有两个原子选择需要进行RMSD计算,比较两个分子中的原子列表。第一个选择的第一个原子与第二个选择的第一个原子进行比较,第五次与第五次,依此类推。实际的顺序与输入PDB文件中的顺序相同。
一旦做出这两个选择,RMSD计算就是调用度量RMSD函数的问题。这里有一个例子: set sel1 [atomselect 0 "backbone"] set sel2 [atomselect 1 "backbone"] measure rmsd $sel1 $sel2 Info) 10.403014
这将打印分子0的主原子与分子1的主原子之间的RMSD。 您还可以在这些计算中使用加权因子。理解如何做到这一点的最好方法是看另一个例子: set weighted_rmsd [measure rmsd $sel1 $sel2 weight mass] Info) 10.403022
在这种情况下,重量是由每个原子的质量决定的。实际上,该术语实际上是原子选择可用的标准关键字之一。其他包括index和ressid(这两个词用起来都很奇怪)以及charge, beta和occupancy。如果您想为权重因子指定自己的值,最后这些项很有用。
计算对齐最适合的对齐分为两个步骤。首先是计算4×4矩阵变换,它将一组坐标变换到另一组坐标。这是用measure fit命令完成的。 假设与之前相同的选择: set transformation_matrix [measure fit $sel1 $sel2] Info) {0.971188 0.00716391 0.238206 -13.2877} {0.0188176 0.994122 -0.106619 3.25415} {-0.23757 0.108029 0.965345 -2.97617} {0.0 0.0 0.0 1.0}
与RMSD计算一样,您还可以在末尾添加一个可选的权重术语。
下一步是使用move命令将矩阵应用于一组原子。到目前为止,你有两个坐标集。您可能认为可以执行类似$sel1 move $transformation matrix的操作,将矩阵应用于该选择的所有原子。你可以,但那不是正确的选择。 需要记住的是,$sel1是主原子的选择。你真的想移动它所连接的整个片段,甚至是整个分子。(这就是前面的讨论发挥作用的地方。)所以你需要做第三个选择,包含所有要移动的原子,然后对这些原子进行变换。 # molecule 0 is the same molecule used for $sel1 set move_sel [atomselect 0 "all"] $move_sel move $transformation_matrix
举一个更复杂的例子,假设你想让分子1和分子9在两个体系中只使用残基4到10的主链原子。方法如下: # compute the transformation matrix set reference_sel [atomselect 9 "backbone and resid 4 to 10"] set comparison_sel [atomselect 1 "backbone and resid 4 to 10"] set transformation_mat [measure fit $comparison_sel $reference_sel] # apply it to all of the molecule 1 set move_sel [atomselect 1 "all"] $move_sel move $transformation_mat
模拟示例脚本这里有一个较长的脚本,您可能会觉得有用。问题是计算模拟的每一帧和第一帧之间的RMSD。通常在模拟中没有初始全局速度,所以质心不会移动,但由于角旋转和数值不精确的缓慢建立,脚本在计算RMSD之前对分子进行对齐。
# Prints the RMSD of the protein atoms between each \timestep # and the first \timestep for the given molecule id (default: top) proc print_rmsd_through_time {{mol top}} { # use frame 0 for the reference set reference [atomselect $mol "protein" frame 0] # the frame being compared set compare [atomselect $mol "protein"] set num_steps [molinfo $mol get numframes] for {set frame 0} {$frame < $num_steps} {incr frame} {
# get the correct frame $compare frame $frame # compute the transformation set trans_mat [measure fit $compare $reference] # do the alignment $compare move $trans_mat # compute the RMSD set rmsd [measure rmsd $compare $reference] # print the RMSD puts "RMSD of $frame is $rmsd" } }
要使用此功能,请加载带有动画的分子(例如,$VMDDIR/proteins/alanin)。来自VMD发行版的DCD)。然后运行print rmsd through time。示例输出如下所示:
vmd > print_rmsd_through_time RMSD of 0 is 0.000000 RMSD of 1 is 1.060704 RMSD of 2 is 0.977208 RMSD of 3 is 0.881330 RMSD of 4 is 0.795466 RMSD of 5 is 0.676938 RMSD of 6 is 0.563725 RMSD of 7 is 0.423108 RMSD of 8 is 0.335384 RMSD of 9 is 0.488800 RMSD of 10 is 0.675662 RMSD of 11 is 0.749352 [...]
|