|
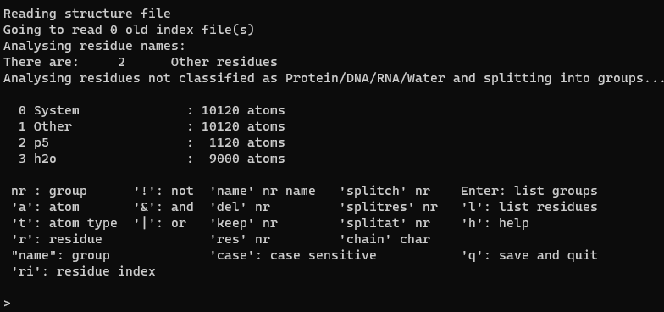
gmx make_ndxmake_ndx程序可用来选择原子组(要分析的某些特定原子或残基的ID标签)并创建索引文件. GROMACS已经定义了一些默认的组, 对普通分析可能够用了, 但如果你想进行更深入的分析, 如为了在模拟中固定某些特定的组, 或获得某些组的特殊能量信息, 则需要使用make_ndx程序来指定这些组. 运行make_ndx后, 可使用r选择残基, a选择原子, name对多组进行改名, 还可以使用|表示或运算, &表示与运算. 下面是几个简单的例子: r 56: 选择56号残基 r 1 36 37: 选择不连续的残基 r 3-45: 选择3至45号残基, 使用连接符指定残基标号范围 r 3-15 | r 23-67: 选择3至15, 23至67号残基 r 3-15 & 4: 选择3至15号残基的主干链原子, 在索引文件中, 4号组为默认的主干链. r 1-36 & a C N CA: 使用包含&的命令指定只包含骨架原子的残基范围 新建索引组的默认名称(如r_1_36_37)很繁琐, 可以使用name命令进行修改. 如name 15 Terminal可将组15的名称改为Terminal. 修改后我们可以使用v命令查看名称是否修改成功, 使用q命令保存修改并退出. 需要注意的一点就是, 对make_ndx的选择, 处理是由左向右依次执行的, &和|没有优先级别之分. 如r 1-3 | r 5-9 & CA会先选择1-3, 5-9号残基, 再从中选择CA原子。 Examples: > 2 | 4 & r 3-5 selects all atoms from group 2 and 4 that have residue numbers 3, 4 or 5 > a C* & !a C CA selects all atoms starting with 'C' but not the atoms 'C' and 'CA' > "protein" & ! "backb" selects all atoms that are in group 'protein' and not in group 'backbone' gmx make_ndx -f npt.gro
git bash无法使用
nr:按数字或带引号的字符串选择索引组。字符串首先与整个组名称相匹配,然后对抗开始,最后对抗任意子字符串。多重匹配是一个错误。 a nr1 [nr2…]:选择原子,原子编号从1开始。 a nr1-nr2:选择从nr1到nr2范围内的原子。 a name1[*] [name2[*]…]:按名称“?”选择原子匹配任何字符,名称末尾允许使用通配符“*”。 t type1[*] [type2[*]…]:作为“a”,但对于类型,需要运行输入文件。 r nr1[ic1] [nr2[ic2]…]:通过数字和插入代码选择残基。 r nr1-nr2:选择从nr1到nr2范围内的残基。 r name1[*] [name2[*]…]:作为“a”,但用于残基名称。 ri nr1-nr2:选择从nr1到nr2范围内的残差索引,1-索引(与数字相反)。 chain ch1[ch2…]:按链标识符选择原子,无法将.gro文件作为输入。 !:取一个群相对于所有输入文件中的原子。 & |:AND和OR,可以放在任意选项之间在上面,输入是从左到右处理的。 name nr name:将组nr重命名为name。 del nr1[-nr2]:删除从nr1到nr2范围内的一个或多个组。 keep nr:删除除nr以外的所有组。 case:使所有名称比较区分大小写。 splitch nr:使用CA距离将组拆分为链。 splitres nr:将基团拆分成残基。 spliat nr:将基团分裂成原子。 res nr:将组中的数字解释为残数 Enter:列出当前定义的组和命令 l:列出残基。 h:显示此帮助。 q:保存并退出。 |