|
gmx trjconv转换和操控轨迹文件
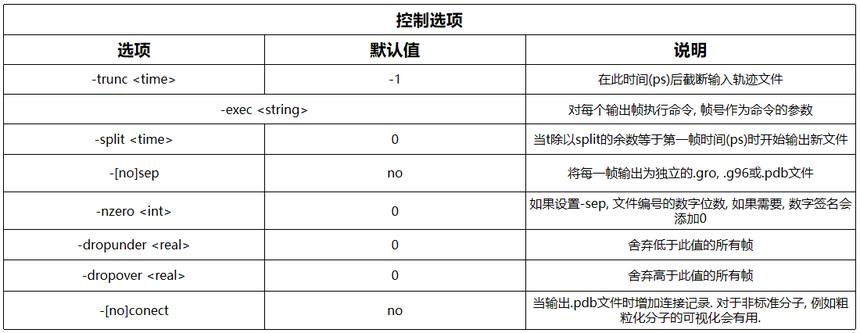
gmx trjconv可以以多种方式来转换轨迹文件: 从一种格式转换为另一种格式 选择一组原子集合 改变周期性的表示方式 将多聚体分子保持一起 将原子在盒子内居中 将原子叠合到参考结构 减少帧数 改变每帧的时间戳(-t0和-timestep) 根据索引文件中的信息把轨迹分割为小的子轨迹, 这样对子轨迹的后续分析就变为对团簇的分析. 需要使用选项-sub. 处理时假定索引文件中的条目为帧数, 并将把索引文件中的每个组输出为单独的轨迹. 选取某个量处于一定范围内的帧, 这个量由.xvg文件给出 gmx trjconv更适用于将多个轨迹文件拼合起来. gmx trjconv支持以下格式的输入和输出文件: .xtc, .trr, .trj, .gro, .g96和.pdb. 文件格式由文件的扩展名决定. 对.xtc, .gro和.pdb输入格式, .xtc和.gro输出文件的精度取决于输入文件; 对其他输入格式, 输出文件的精度由-ndec选项决定. 如果设定了-ndec选项, 输出格式的精度总是取决于-ndec. 所有其他格式的精度都是固定的. .trr和.trj输出格式的精度可以是单精度或双精度, 取决于gmx trjconv程序的精度. 注意, 只有.trr, .trj, .gro和.g96格式的文件支持速度. -sep选项可将每一帧写入到单独的.gro, .g96或.pdb文件. 默认情况下, 所有帧都被写入到一个文件中. 拼合了所有帧的.pdb文件可以使用rasmol –nmrpdb来查看. 为了节省磁盘空间, 可以选择部分轨迹并将其写入到一个新的轨迹文件中. 例如, 去除蛋白质水溶液轨迹中的水分子. 始终 要保存原始的轨迹文件! 我们推荐使用可移植的.xtc格式进行分析以节省磁盘空间并得到可移植的文件. 有两个选项可用于将轨迹叠合到参考结构或进行主成分动力学分析. 第一个选项仅仅将结构简单地叠合到结构文件中的参考结构. 第二个选项是逐步叠合: 第一时间步的结构叠合到结构文件中的参考结构, 后续时间步的结构则叠合到前一步的叠合结构. 与常规的叠合方法不同, 利用这种方式可以产生连续的轨迹, 例如当蛋白质的构象转变很大时. 选项-pbc用于设置周期性边界条件的处理方式: mol: 将分子的质心置于盒子中, 需要使用-s提供一个运行输入文件 res: 将残基的质心置于盒子中 atom: 将所有原子置于盒子中 nojump: 检查原子是否跳过了盒子边缘, 如果是则将它们放回来. 这样所有分子都可以保持完整(如果在初始构型中它们是完整的). 注意, 这样可以确保轨迹连续, 但分子可能扩散出盒子. 如果提供了结构文件, 此过程的起始构型将来自结构文件, 否则将使用第一帧的构型. cluster: 将选定索引中的所有原子团簇化, 这样它们到团簇质心的距离最近, 团簇质心会迭代更新. 注意, 只有确实存在一个团簇时, 这种方法才能给出有意义的结果. 幸运的是你以后可以使用轨迹查看器来检查是否存在一个团簇. 同时也要注意, 如果分子破碎了, 这个选项也不会起作用. 单独的选项-clustrcenter可用于指定团簇的近似质心. 这适用于一些情况, 例如存在两个大囊泡, 你需要保持他们的相对位置. whole: 将破碎的分子恢复完整 对-pbc的mol, res和atom选项, -ur选项设置单元晶胞的表示方式. 对于三斜盒子这三个选项会给出不同的结果, 而对于长方盒子, 给出的结构相同. rect是普通的长方体形状, tric是三斜晶胞, compact将所有原子置于离盒子中心距离最近的位置. 这有利于, 例如对截断八面体和菱形十二面体的可视化. 对tric和compact选项的中心是tric(见下文), 除非选项-boxcenter设置了不同的值. 选项-center将体系在盒子内居中, 用户可以选择用于确定几何中心的组. 对-pbc和-center选项, -boxcenter设置盒子的中心位置. 中心的选项为: tric: 盒向量总和的一半, rect: 盒子对角线的一半, zero: 0. 如果居中后你想使所有分子都处于盒子中, 可以使用-center和-pbc mol选项. 选项-box设置新盒子的大小. 此选项只用于主维度, 因此通常只用于长方盒子. 如果你只想修改某些维度, 例如读取轨迹时, 你可以使用-1使某一维度保持不变. 当仅调用一次gmx trjconv时, 使用-pbc, -fit, -ur和-center选项的组合并不总能精确地达到你的目的. 这种情况下可考虑使用多次调用, 可参考GROMACS网站的一些建议. 使用-dt选项可以减少输出中的帧数. 此选项依赖于输入轨迹中时间的精确度, 因此, 如果它们不够精确, 可以使用-timestep选项来修改时间(可以同时进行). 为了制作平滑的电影, gmx filter程序可以使用低通频率滤波器来减少帧的数目, 从而减少了高频运动的走样. 使用-trunc选项, gmx trjconv可以就地截断.trj文件, 即不需要复制文件. 当在磁盘I/O过程中运行崩溃时(即磁盘已满), 或者当拼合两个邻近的轨迹但不能重帧时, 此选项很有用. 选项-dump用于从你的轨迹文件中抽取处于或邻近指定时间的帧. 选项-drop读取.xvg文件中的时间和数值. 当设置了选项-dropunder和/或-dropover, 不会输出低于或高于相应选项设定值的帧.
补充说明 gmx trjconv可能是最常用的后处理工具, 用来处理坐标, 处理周期性或者手动调整轨迹. 利用它抽取特定的轨迹比较简单, 但使用它处理轨迹的周期性时, 一些选项不容易理解. 下面对其中的一些进行说明. -pbc mol|res|atom指定以何种方式考虑PBC, 是使分子的质心, 残基的质心, 还是每个原子处于盒子中. 如果使用-pbc atom所有原子都处于盒子之中, 这样边界上的分子看起来破碎了. 如果对破碎后的分子再使用一次-pbc whole, 将分子恢复完整, 其效果与-pbc mol类似. -pbc nojump可以保证分子的运动是连续的, 就像体系处于真空中一样, 分子连续地向各个方向扩散. 在计算MSD这样的量的时候, 需要这样考虑. 但gmx msd在计算时已经考虑了这点, 所以我们就无须先利用此选项对轨迹进行处理了. 此选项对单个构型没有意义. 使用-pbc mol|res|atom选项时, 会使相应的中心处于盒子中, 而盒子的显示方法则使用-ur来控制. 如果使用长方体盒子, -ur的三种选项给出的结果相同, 所以无需考虑此项. 如果使用了三斜盒子, -ur的三种选项给出的结果不同: -ur tric粒子处于三斜盒子中, -ur rect粒子处于长方盒子中, -ur compact粒子处于距盒子中心最近的位置, 近似球形. 利用-center选项可使某组原子在盒子内居中, 运行时, 会提示你选择要居中的组. 此选项可以和-pbc mol|res|atom一起使用, 达到使某组原子居中, 同时其他原子都处于盒子内的目的. 在使用-ur tric|compact, -pbc mol|res|atom|, -center选项时, 都需要定义盒子的中心. 默认使用的盒子中心处于盒向量的一半处. 但可以使用-boxcenter改变: tric盒向量总和的一半, rect盒子对角线的一半, zero0. 上面的这几个选项可组合使用, 但不能保证一定能满足需要, 有时可能需要使用gmx trjconv多次. 注意, -pbc和-fit rot两个选项不能一起使用. 否则程序运行错误, 给出如下信息: PBC condition treatment does not work together with rotational fit. Please do the PBC condition treatment first and then run trjconv in a second step for the rotational fit. First doing the rotational fit and then doing the PBC treatment gives incorrect results! 这意味着凡同时涉及周期性和叠合的处理都需要分两次进行, 而且必须先进行周期性处理, 再进行叠合, 否则结果错误. -pbc几种处理的效果可参看 gmx trjconv选项测试 |
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||